|
|
What is a
gene ? Helen Pearson (Nature, vol 441,
25 May 2006, 399-401).
Qu'est-ce qu'un
gène ?
une
traduction
personnelle
de cet article est disponible
ICI.
|
|||||||
retour accueil, nouveau cours 1èreS, plan du cours 1èreS
Plan |
|||||||||
|
|
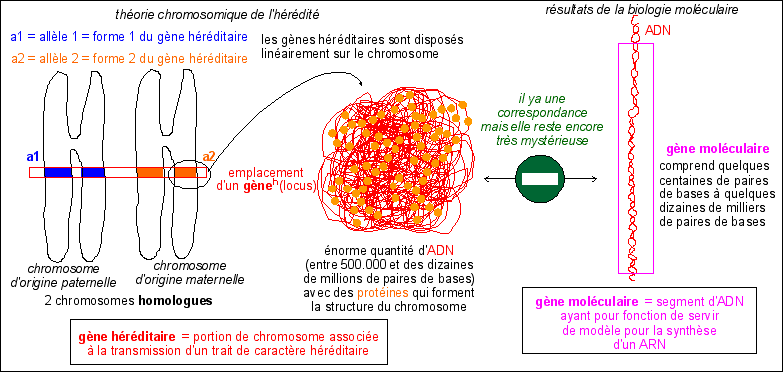
1 -Les acides nucléiques (ADN et ARN) sont de longs polymères de nucléotides 2 - Les protéines sont de longs polymères d'acides aminés 3. L'information génétique stable est contenue dans l'ADN et copiée dans l'ARN lors de la transcription 4 . L'information génétique transitoire des ARNm est traduite en polypeptides (à l'aide d'autres ARN) dans les ribosomes du cytoplasme 5. Les peptides exportés subissent une maturation dans le REL puis dans l'appareil de Golgi avant d'atteindre la membrane ou d'être sécrétés par exocytose 6. Les gènes ne représentent qu'une petite partie de l'ADN des eucaryotes |
||||||||
|
autres pages connexes:
l'alcaptonurie, |
Annexes : - pour en finir avec les confusions nées de l'emploi des termes génotype, phénotype, allèle et gène dans le sens héréditaire, chez les procaryotes et les eucaryotes - pages sur les génomes (en travaux) |
||||||||
|
Remarque de vocabulaire: |
|
|
|||||||||||||||||||||||||||||||||||||
|
1 -Les
acides nucléiques (ADN et ARN) sont de longs
polymères de nucléotides
|
|
|
|||||||||||||||||||||||||||||||||||||
|
|
|
Les acides nucléiques sont des polymères (du grec poly = plusieurs et merein = partage) (voir cours de seconde) car il sont composés d'unités identiques (les monomères). Comme il peut y avoir plusieurs types de nucléotides on parle de copolymères. |
|
Les nucléotides sont des molécules composées d'une base azotée (A= adénine, T=thymine, U=uracile, C=cytosine, G=guanine), d'un sucre (ribose ou désoxyribose) et d'un, deux ou trois groupement(s) phosphate(s). Les nucléotides ne sont pas que des composants des acides nucléiques mais ont des fonctions variées dans la cellule: par exemple l'ATP (adénosine triphosphate = adénine + ribose (l'adénosine est le nom du nucléoside) + 3 phosphates) et ses dérivés monophosphate (AMP) ou diphosphate (ADP) qui sont des molécules énergétiques intermédiaires (transfert de l'énergie chimique de liaison entre molécules) mais aussi informatives (AMP cyclique ou cAMP par exemple); ou le dGTP (adénine+désoxyribose+3 groupements phosphate; le "d" devant la molécule indiquant la nature du sucre: le désoxyribose) qui est aussi une molécule énergétique. |
|||||||||||||||||||||||||||||||||||
|
TD
- ADN-ARN (les acides
nucléiques);
forme, structure des molécules; simualisation*
3D (attention
applet Jmol de 540Ko à télécharger) |
|
1.2 - L'ADN est une longue molécule en hélice à deux brins L'ADN (acide
désoxyribonucléique) est formé de
deux chaînes (brins) de nucléotides
dessinant une double hélice dont le
diamètre est de 2 nm. |
|||||||||||||||||||||||||||||||||||||
|
|
Les ARN (acides
ribonucléiques) sont des chaînes (un seul
brin) de nucléotides pouvant se replier et s'apparier
partiellement. |
||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||
|
Il existe 20 acides aminés dans les protéines du vivant. Ce sont des molécules organiques donc composées de C, H, O, N et parfois S. Elles ont toutes deux groupes d'atomes que l'on appelle des radicaux et qui leur donnent leur nom: un radical acide (-COOH) et un radical amine (-NH2). |
|
Les protéines sont des peptides dont le poids moléculaire est supérieur à 10.000 daltons (un dalton est le poids d'un atome d'hydrogène). |
|
On utilisera souvent en lycée le mot protéine comme équivalent de peptide (voir remarque ci-dessus). |
|||||||||||||||||||||||||||||||||||
|
TP-TD
- Acides aminés et
protéines;
simualisation* en
3D
(attention applet
Jmol de 540Ko à télécharger) |
|
Les protéines sont souvent formées de l'assemblage de plusieurs molécules et contiennent des éléments non peptidiques (appelés groupement prosthétique, comme le groupe tétrapyrolique à Fe qui forme l'hème et s'ajoute à la molécule de globine de l'hémoglobine... elle-même formée de 4 sous-unités identiques 2 à 2). |
|||||||||||||||||||||||||||||||||||||
|
3. L'information génétique stable est contenue dans l'ADN et copiée dans l'ARN lors de la transcription |
|||||||||||||||||||||||||||||||||||||||
|
|
|
|
|
3.1 - La séquence des
molécules polymériques constitue une
information linéaire
|
|||||||||||||||||||||||||||||||||||
|
|
|
La suite linéaire (en ligne) des nucléotides d'une chaîne de l'ADN (l'autre étant déterminée par complémentarité des bases), ou de l'unique chaîne des ARN, forme une séquence propre à chaque molécule d'acide nucléique. Elle peut être représentée par la suite des bases (par exemple .....AACTAGGCTTAA....). |
|
De même la suite linéaire des aa d'une protéine est appelée séquence ou structure primaire. |
|
Les séquences constituent une information linéaire qui n'est pas sans rappeler l'information exprimée par la suite de lettres d'un mot. Mais ce n'est pas la seule information que peut donner une molécule qui se déploie dans l'espace avec une forme. Même si la forme est indubitablement liée à la séquence elle n'en est pas la résultante car la forme dépend aussi des conditions de milieu et de molécules additionnelles pouvant s'ajouter pour donner une structure complexe. Par exemple l'ADN s'associe avec des protéines pour donner un chromosome chez les eucaryotes et les protéines sont nombreuses à s'associer entre elles ou avec des ARN pour donner des complexes. |
|||||||||||||||||||||||||||||||||
|
|
|
|
|
|
|||||||||||||||||||||||||||||||||||
|
La transcription est la copie de l'un des brins de l'ADN en ARN. |
|
C'est donc la synthèse d'une molécule d'ARN (monobrin) à partir d'un l'un des brins d'une molécule d'ADN (brin matrice servant à une copie complémentaire base à base). |
|
Lors de la transcription, la molécule d'ADN est maintenue ouverte (brins séparés) par un gros complexe enzymatique : l'ARN polymérase (ARN polymérase ADN dépendante qui contient du Zn2+) dans une zone d'une longueur de 17 paires de bases (3,4 nm pour 10 pb) ce qui nécessite, du fait de l'enroulement de l'ADN, de dérouler l'ADN en amont et de le réenrouler en aval de la zone de transcription. L'ARN polymérase copie un des brins de l'ADN (brin non codant ou brin ADN matrice ou brin transcrit) en un ARN complémentaire à une vitesse qui atteint 50 nucléotides à la seconde chez E. coli. La transcription nécessite l'ion Mg2+ et de l'énergie fournie par les ribonucléosides triphosphate (ATP, GTP, CTP et UTP). |
|
Toutes les étapes de la transcription sont régulées par des enzymes ou des facteurs par de nombreux mécanismes qui constituent le cœur de la régulation de l'expression génétique.
|
|||||||||||||||||||||||||||||||||
|
|
|
|
|
|
|||||||||||||||||||||||||||||||||||
|
|
|
Il existe plusieurs types d'ARN. Pour simplifier on ne parlera que des ARN messagers et des "autres ARN" pour désigner les ARN qui ne sont pas traduits. |
|
Si l'ensemble des gènes moléculaires constitue le génome, les gènes moléculaires qui sont transcrits en ARNm (qui seront traduits à leur tour en protéines) constituent le protéome. Les gènes moléculaires transcrits mais par forcément traduits constituent le transcriptome. |
|
Remarque: |
|||||||||||||||||||||||||||||||||
|
|
|
|
|
|
|||||||||||||||||||||||||||||||||||
|
|
|
||||||||||||||||||||||||||||||||||||||
|
Remarques: |
|
1 - L'ADN des mitochondries et des chloroplastes est transcrit directement au sein de ces organites. 2 - Les ARNm subissent de nombreuses maturations avant d'être éventuellement traduits. 3 - L'ensemble des ARNm d'une cellule forme le transcriptome; pour un fibroblaste humain par exemple il peut comprendre jusqu'à 300.000 molécules d'ARNm (une cellule humaine comprenant 50.000 gènes moléculaires environ mais on pense que seuls 10.000 à 20.000 d'entre eux sont actifs dans une cellule différenciée), chaque ARNm n'étant présent que sous la forme d'au maximum 15 copies. On sait détecter par la technologie des puces à ADN (DNA chips) une unique copie d'un ARNm dans un broyât cellulaire... |
|
4 - L'information génétique contenue dans l'ARN peut passer dans l'ADN par transcription inverse grâce à des molécules de type ADNpolymérase ARNdépendante (ou transcriptase inverse): voir le cours d'immunologie de TS sur le VIH. Depuis les années 80-90 où on les recherche, on a trouvé des transcriptases inverses chez des bactéries, des mycètes et même chez les ovocytes de poissons. 5 - L'ADN peut être copié (répliqué) par des enzymes (ADN polymérase). L'ARN peut aussi être répliqué par des ARN polymérases. Au total on a donc 4 types de synthèses d'acides nucléiques: 2 réplications, isosynthèses à partir d'un brin de même nature que le brin synthétisé, et 2 hétérosynthèses à partir d'un brin matrice de nature différente. |
|
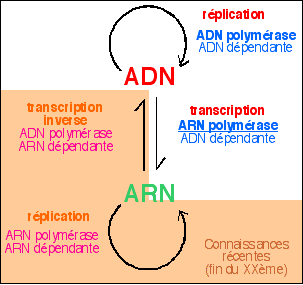 On notera cependant que les complexes enzymatiques ARN dépendants (en rose) n'ont pas été trouvés dans toutes les cellules et que ces voies ne sont pas généralisables. Le fait qu'elles existent ouvre la voie à une nouvelle compréhension de la notion d'information génétique. |
|||||||||||||||||||||||||||||||||
|
|
|
||||||||||||||||||||||||||||||||||||||
|
L'ARN doit être considéré comme faisant partie intégrante de l'information génétique. |
|
Les découvertes datant de la fin
du XXème siècle remettent en cause le dogme
central de la biologie moléculaire d'alors selon
lequel l'information génétique est contenue
dans le seul ADN est exprimée dans l'ARN. L'ADN a
été longtemps considérée comme
la seule information génétique à
caractère héréditaire étant
donné qu'elle était la seule stable. Mais on a
commencé à reconsidérer le statut des
ARN depuis que son information peut être transmise
à la cellule sous forme d'ADN par transcription
inverse. On a de fait trouvé des transcriptases
inverses dans de nombreuses cellules procaryotes et
eucaryotes (voir travaux
de Beljanski, du point de vue
historique, sur une ancienne page d'histoire de la
génétique...). |
|
L'article de S. Lolle et R. Pruitt met en
lumière le fait considéré comme de plus
en plus courant qu'il existe des réversions de
certains gènes associés à des
protéines. Une réversion apparemment
dirigée ou paramutation
consiste chez un individu présentant un gène
muté identifié, à
récupérer (par un procédé
inconnu mais les auteurs proposent l'hypothèse d'une
matrice d'ARN transmise par une
hérédité non mendélienne...), au
bout de quelques générations, une ou deux
copies du gène ancestral non muté. La
fréquence des réversions peut atteindre
quelques dizaines de % des descendants. Ici, ces chercheurs
ont travaillé sur le modèle
génétique usuel qui est une petite plante:
Arabidopsis thaliana, à partir d'un
gène HOTHEAD (HTH) associé à une
protéine ACE (ADHESION OF CALYX EDGES) de 594 aa
intervenant dans la signalisation cellulaire et
située pour une bonne part vers l'extérieur de
la cellule; cette protéine possède une
activité enzymatique (de type mandelonitrile lyase
qui semble être une propriété
très générale de nombreuses
protéines extracellulaires... voir La Recherche, 386,
mai 2005, pp14-15). Le gène HTH associé est
exprimé dans les fruits, les racines,
l'inflorescence, les fleurs, la feuille et la tige; on,
connaît une impressionnante liste de 22 mutants dont
ceux étudiés par cette équipe. La quasi
intégralité (il manque au moins quelques
figures...) de l'article en anglais peut être
consultée à l'adresse: http://www.euchromatin.com/Pruitt01.htm
mais il ne peut s'adresser qu'à des enseignants de
SVT ayant un bon niveau de génétique et je ne
pense pas qu'il apporte quelque lumière
supplémentaire. Il peut être
complété par les données du site
http://www.arabidopsis.org/
pour les gènes clonés et leurs produits.
|
|||||||||||||||||||||||||||||||||||
|
pages
sur les
génomes
Voir la traduction de l'article d'Helen Pearson: Qu'est-ce qu'un gène ? |
|
|
|||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||
|
|
|
Les ribosomes sont de petits (20 nm environ, un peu plus petits chez les procaryotes, un peu plus gros chez les eucaryotes) complexes ARN-protéines composés de 2 sous-unités qui s'assemblent lors de la traduction. Les ribosomes sont associés
à un ARNm en polysomes. Un polysome se
présente au MET comme une chaîne
(parfois fixés au MET sous la
forme d'une spirale) de ribosomes le
long d'une molécule d'ARNm qu'ils traduisent. |
|
Lors de la traduction les aa sont présentés aux ribosomes sous une forme activée (qui nécessite de l'ATP) et associés à des ARN transporteurs-adaptateurs (de transfert = ARNt). Ils sont liés progressivement les uns aux autres par des liaisons covalentes (peptidiques) dans l'ordre déterminé par l'ARNm. En effet, chaque triplet de base de l'ARNm (codon) est associé à un aa activé grâce à son ARNt transporteur-adaptateur spécifique. La correspondance entre les codons de l'ARNm et les aa constitue le code génétique. |
|
Le code génétique est
universel (toutes les systèmes de traduction
cellulaire utilisent les mêmes correspondances
codon-aa). Cette universalité n'est pas absolue mais
la rareté des exceptions (par exemple dans les
mitochondries) prouve au contraire que ces exceptions sont
plutôt des optimisations que des dérogations
à la règle (il n'y a
qu'une dizaine d'aa dans les peptides traduits dans la
matrice mitochondriale et les ARNt mitochondriaux sont issus
du génome mitochondrial)
prouve au contraire l'importance de l'universalité du
code: tout changement a des conséquences très
graves pour le sens de l'information génétique
de la cellule. |
|||||||||||||||||||||||||||||||||
|
Remarque: Les mitochondries et les chloroplastes possèdent des ribosomes qui réalisent la traduction des ARNm directement dans leur compartiment matriciel. De nombreuses protéines des ces compartiments possèdent des sous-unités codées à la fois par le génome nucléaire et le génome mitochondrial ou chloroplastique. |
|
Les ribosomes lient les aa correspondant à deux codons successifs puis progressent le long de l'ARNm en se décalant d'un codon. La traduction comporte un phase d'initiation (codon initiateur AUG correspondant à la Met), une phase d'élongation (20 aa par seconde environ), et une phase de terminaison (codon stop: UGA, UAA, UAG). La traduction est un mécanisme métabolique qui consomme de l'énergie (GTP) et qui est contrôlé par de nombreuses enzymes. |
|
Les ARNm sont traduits plusieurs fois avant d'être détruits. |
|
La plupart des protéines sont organisées en complexes fonctionnels qui s'auto-assemblent sous le contrôle d'enzymes et de facteurs spécifiques (par exemple les molécules chaperons qui assurent le repliement de certaines protéines...). |
|||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||
|
|
|
||||||||||||||||||||||||||||||||||||||
|
Les enzymes doivent être étudiées autre part dans le cours mais il est probablement judicieux de présenter la transcription comme une réaction enzymatique Un exemple d'enzyme particulièrement simple à comprendre est celui des ribosomes. Ces particules sont maintenant très bien connues et l'on peut affirmer que d'une part ce sont bien leur propriétés topologiques qui leur confèrent leur fonction et d'autre part que ces complexes ARN-protéines sont restitués intégralement sans altération en fin de réaction (ce que l'on appelle le cycle ribosomial): ces deux propriétés en font des enzymes au sens général. À ce sujet on dit souvent que les ribosomes sont les catalyseurs de la synthèse protéique, ce qui correspond bien à les apparenter à des enzymes. |
|
La synthèse des protéines, une
réaction
enzymatique catalysée par les
ribosomes, l'ARNm, les ARNt et de nombreux cofacteurs de
traduction |
|||||||||||||||||||||||||||||||||||||
|
|
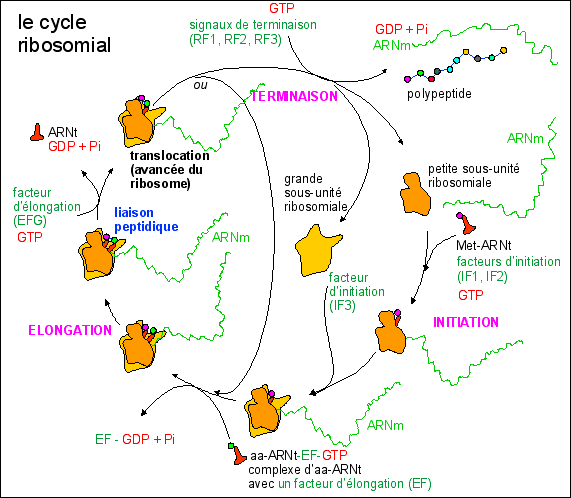 le GTP, guanosine triphosphate,
est un ribonucléotide
énergétique tout comme l'ATP,
adénosine triphosphate qui est le
ribonucléotide le plus courant dans la
cellule; la méthionine (Met) est le premier
acide aminé (aa) de toute chaîne
polypeptidique (qui comporte n aa ici). C'est un
domaine particulier de la grande sous-unité
ribosomiale (la peptidyl-transférase)
qui catalyse l'établissement de la liaison
peptidique. On notera que le ribosome (avec ses 3
(chez les procaryotes) ou 4 (chez les eucaryotes)
molécules d'ARNr; et ses quelques 53 (82)
protéines) possède des sites de
fixation de tous les cofacteurs, de l'ARNm et
des complexes aa-ARNt.
|
||||||||||||||||||||||||||||||||||||||
|
exemple détaillé dans la page sur les enzymes |
|
|
|||||||||||||||||||||||||||||||||||||
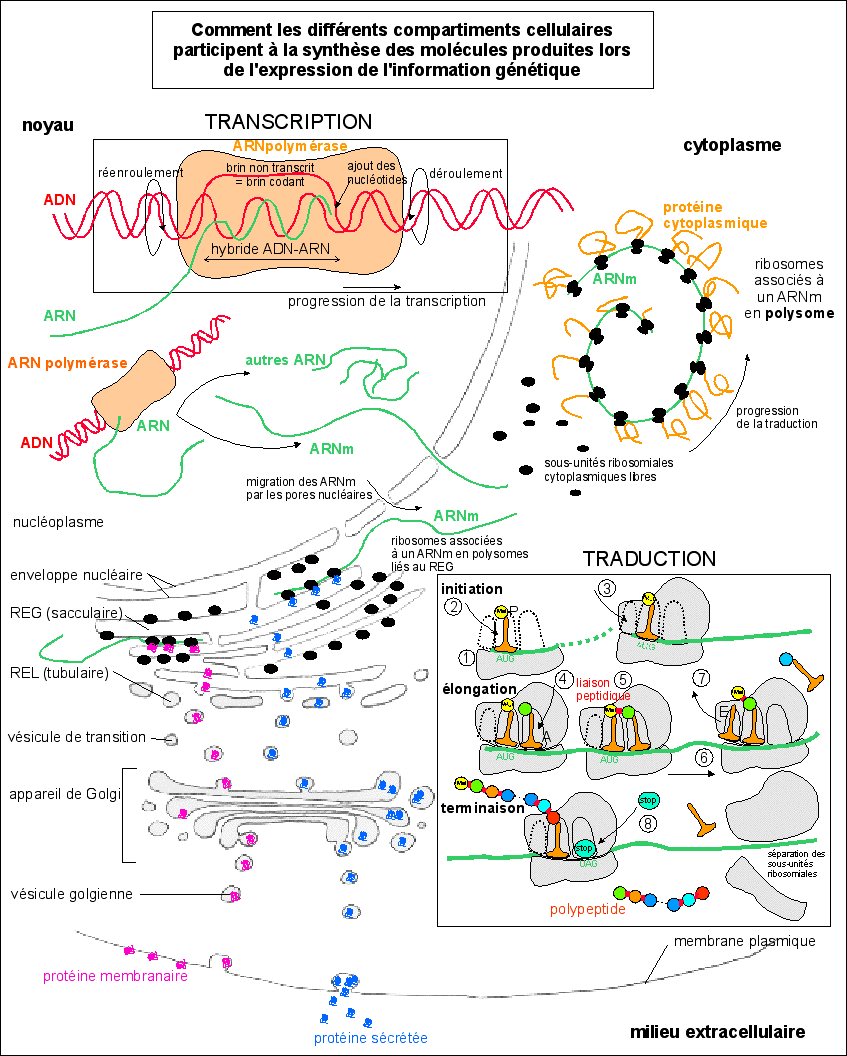 Les étapes de la transcription sont légèrement plus détaillées que l'exige le strict respect du programme: cependant seuls les ARNt formant des complexes avec les aa transportés sont représentés, ce qui me paraît indispensable à la compréhension de la traduction; pour éviter toute polémique stérile vous pouvez les appeler "ARN adaptateur-transporteur d'aa". Les étapes de la traduction sont les suivantes: 1 - assemblage des 2 sous-unités ribosomiales autour de l'ARNm; 2 - arrivée et mise en place d'un aa (transporté par son ARN adaptateur-transporteur) qui correspond selon le code génétique au codon de l'ARNm placé à la base du premier site de fixation des ARN transporteurs-adaptateurs dans le ribosome; on notera que le premier aa est toujours une méthionine (Met) car le premier codon (initiateur) est toujours AUG; 3 - arrivée d'un autre aa transporté par un ARN adaptateur-transporteur dans le second site ribosomial; 4 - établissement d'une liaison peptidique entre les deux aa accolés; 5 - déplacement du ribosome d'un codon le long de l'ARNm; 6 - ce qui implique le départ de l'ARN transporteur adaptateur qui se détache du peptide en cours de formation et un changement de site ribosomial pour l'autre ARN transporteur-adaptateur qui porte la chaîne peptidique en formation; 7 - après de nombreux cycles d'élongation, lorsque un codon STOP (UAG, UGA, UAA) devient accessible dans le second site ribosomial, un complexe vient se fixer sur ce codon et provoque la terminaison de la traduction, c'est-à-dire la libération de la chaîne peptidique et de l'ARN transporteur-adaptateur et la séparation des sous-unités ribosomiales. |
|||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||
|
|
|
Les peptides synthétisés dans le cytoplasme sont directement utilisés par la cellule pour le métabolisme (dans des enzymes...) ou pour des structures (histones...) (voir tableau). |
|
Les peptides synthétisés à la surface du REG migrent dans le REL (reticulum endoplasmique lisse, qui est tubulaire) puis dans l'appareil de Golgi et sont exportés dans des vésicules soit vers le membrane (protéines membranaires) soit vers l'extérieur de la cellule (protéines sécrétées par exocytose). |
|
C'est par exemple dans l'appareil de Golgi que sont accrochés les sucres (résidus glucidiques) des glycoprotéines. |
|||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||
|
Un gène moléculaire est défini par sa nature chimique (ADN), sa séquence (suite de monomères) et sa fonction (servir de copie lors de la transcription). |
|
Si l'on considère que les gènes moléculaires contiennent une information, cette information est linéaire. De plus, comme la seule fonction connue précisément de l'ADN, découpé en gène moléculaires, est une fonction passive: servir de modèle lors de la transcription, l'information génétique est donc une information pour une molécule (d'ARN). |
|
L'expression de l'information
génétique stable de l'ADN passe d'abord par
l'ARN puis peut passer par un polypeptide. |
|
Les
gènes
moléculaires sont donc des
segments
d'ADN contenant une
information
linéaire
exprimée dans une
molécule de type ARN lors de la
transcription
puis, le plus souvent, dans une molécule peptidique
lors de la
traduction. Le contrôle de l'expression de l'information génétique peut se faire au niveau de la transcription ou de la traduction. |
|||||||||||||||||||||||||||||||||
|
pages
sur les
génomes |
|
Le nombre de gènes moléculaires des différentes cellules vivantes n'est pas précisément connu. Je vous rappelle que séquencer la totalité de l'ADN d'une cellule ne veut par dire séquencer le génome (ensemble des gènes) car on n'est pas du tout sûr que tout l'ADN (des eucaryotes surtout) est organisé en gènes moléculaires. On estime à 2000 ou 4000 le nombre de gènes moléculaires d'Escherichia coli, 6340 celui de Saccharomyces cerevisiae, 10.000 celui des cellules de Drosophila melanogaster et peut-être 100.000 ou 50.000 pour l'homme. |
|
Un gène moléculaire peut correspondre à plusieurs produits et inversement un produit peut nécessiter plusieurs gènes moléculaires, les gènes moléculaires peuvent se chevaucher... bref, ceci est une autre histoire: celle de la génomique (science des gènomes) ou biologie moléculaire du gène moléculaire (avec sa nouvelle branche: la protéomique qui s'intéresse aux seuls gènes moléculaires contenant une information pour des protéines) et elle ne fait que commencer (voir histoire de la génétique). |
|
Malgré le séquençage complet du génome humain on n'espère au mieux que 25.000 gènes moléculaires associés à des protéines. Si on ajoute les quelques 2.000 gènes moléculaires associés à des ARN non traduits cela fait un peu court pour que l'imaginaire continue de voir dans les protéines autre chose qu'un support matériel de la vie. Quand au "pouvoir magique" du gène moléculaire il s'estompe petit à petit. |
|||||||||||||||||||||||||||||||||
|
Remarque: |
|
Certains bioinformaticiens, cherchant à chiffrer le niveau de complexité du vivant, considèrent qu'il y a encore 7 ordres de grandeur à atteindre avant de pouvoir rendre compte du niveau cellulaire (génomique comparative, interactions rigides, génome, métabolisme et signalisation unicellulaire, interactions flexibles, complexes protéiques, métabolisme et signalisation pluricellulaires, interactions des complexes protéiques). À mon avis le problème est pris à "rebrousse poil". La vie, dans sa complexité, peut être supportée par des formes simples dans des espaces de contrôle multidimensionnels. Il semble nécessaire de cesser de raisonner dans un espace à deux dimensions avec une infinité de formes. La théorie des modèles (géométrique) de René Thom permet de complexifier l'espace pour simplifier les formes. Ce qui est moins intuitif mais très compréhensible au moins jusqu'à la dimension 4. |
|
|
|||||||||||||||||||||||||||||||||||
|
|
|
Le génome humain
(au sens large de la totalité
de l'ADN nucléaire d'une cellule
humaine) de 3,1 milliards de paires
de bases contiendrait 20.000 à 25.000 gènes
protéiques....(Pour la
Science, dossier n°46, 2005, p 18)
|
|
|
|||||||||||||||||||||||||||||||||||
|
|
|
|
** l'ADN satellite sont des régions répétées des millions de fois de quelques centaines de paires de nucléotides; elles sont souvent localisées près du centromère du chromosome mais peuvent occuper un bras entier. Ces séquences sont variables entre individus, même d'espèces voisines. Si l'ADN satellite représente 6,5% de l'ADN humain, 50% de l'ADN humain est répété. Les éléments transposables représentent ainsi 44 % de l'ADN ; on y trouve pour 3% des transposons*** (fragments d'ADN qui se déplacent directement dans l'ADN en s'intégrant ou en se coupant), pour 8 % des éléments possédant des LTR (longues répétitions terminales) aux deux extrémités et qui se comportent comme des rétrovirus (car ce sont des séquences d'ADN qui contiennent le gène d'une transcriptase inverse et qui se déplacent dans l'ADN par l'intermédiaire d'une forme ARN retrotranscrite) - on les qualifie de rétrovirus endogènes**** - et pour 33% d'autres éléments qui contiennent aussi une transcriptase inverse. La plupart des élément transposables sont non fonctionnels. On pense que ce sont les vestiges de gènes ou de séquences qui se sont déplacés chez des ancêtres plus ou moins lointains. |
||||||||||||||||||||||||||||||||||||
|
|
|
|
|||||||||||||||||||||||||||||||||||||
|
Il existe un
deuxième niveau de
compréhension de la fonction du gène
moléculaire car les
gènes ne sont pas juxtaposés, figés et
isolés mais sont emboîtés
(plusieurs produits peuvent être
synthétisés à partir d'un seul
gène selon l'environnement), dynamiques (le
début et la fin du gène peuvent varier, le
génome d'une cellule évolue avec son
âge...), et interdépendants
(l'expression d'un gène est sous le contrôle
d'autres gènes et souvent sous le contrôle de
ses produits (rétrocontrôle)...). Mais cette
complexité, qui fait appel notamment à la
science des réseaux, dépasse de loin le niveau
du lycée. |
|||||||||||||||||||||||||||||||||||||||
|
|
|
|
|||||||||||||||||||||||||||||||||||||