Qu'est-ce que le code génétique ?
Le code génétique a une logique
|
Le code génétique est un tableau (dictionnaire de traduction) des correspondances entre les 64 triplets de bases azotées de l'ARNm (codons) et les acides aminés.
|
Il y a 64 codons: Les anti-codons sont les ensembles de 3 lettres de l'ARNt complémentaires de l'ARNm. Il y a 20 acides aminés "standard" reconnus par les ARNt (soit 32 ARNt standard). |
Sources: Principes de Biochimie, Lehninger, Nelson et Cox, 1994, Flammarion, 5-1 Pour la Science, Dossier n°46, janvier-mars 2005, p 15, figure 5 |
||||
|
N.B.1 - Ne pas confondre "code génétique" avec "information génétique". L'information génétique est une notion vague et théorique qui signifie les données enregistrées dans les acides nucléiques et les protéines par la cellule: voir cours de 1èreS). N.B.2 - Ce code est le seul qui doive être appelé "code génétique". Toute autre correspondance de bases de l'ADN par exemple avec celles de l'ARN ou, de façon indirecte entre les bases de l'ADN et les acides aminés.... ne doivent pas être appelées ainsi. Remarque: pour ceux qui seraient tentés de pousser un peu loin la métaphore (et non l'analogie) de la séquence de la traduction de l'ADN avec la lecture, je conseille un article de fond de Guiseppe Longo : Giuseppe LONGO, L'alphabet, la Machine et l'ADN : l'incomplétude causale de la théorie de la programmation en biologie moléculaire, présenté lors du Colloque de Philosophie des sciences : LE LOGIQUE ET LE BIOLOGIQUE, 22 avril 2005, Université Paris 1 (Panthéon-Sorbonne). |
||||||
|
Les phénomènes de lecture non-conventionnelle lors de la traduction sont qualifiés de recodage. Pour les esprits curieux je conseille une conférence sur le sujet à l'ENS : "Le recodage : une lecture alternative du code génétique" par Olivier Namy (univ. Paris XI) [16th February, 2005 at 14:30] - http://diffusion.ens.fr /en/ index.php? res=conf&idconf=556) |
C'est au cours des années 1960-1965 que le code génétique fût élucidé (M. Nirenberg, H. Matthaei, P. Leder, H. Gobind Khorana en furent les artisans) même si le tableau ci-dessus n'a été publié dans son intégralité qu'en 1966. |
Ce code est universel. En effet, les rares exceptions que l'on connaît chez quelques bactéries, et quelques eucaryotes, notamment au niveau des mitochondries (qui ont leurs propres ARNt et dont le génome ne code que pour 10 à 20 protéines) et chez quelques unicellulaires, sont regardées plutôt comme des preuves de l'universalité plutôt que comme des exceptions à une règle. On préfère parler de flexibilité contrôlée. Cette universalité est un argument très fort pour l'unité du vivant plus précisément pour une origine commune des mécanismes moléculaires du vivant. |
Ce code est dit
dégénéré (on dit parfois
redondant) ce qui signifie qu'un acide aminé
peut être désigné par plusieurs codons
(seuls la méthionine et le tryptophane n'ont qu'un
seul codon). (Chez E. coli 21 aa
peuvent être reconnus car le codon UGA servant
occasionnellement pour la
sélénocystéine, ce qui rend dans ce cas
le code ambigu.)
Pour des
lecteurs qui maîtrisent l'anglais et qui
désireraient voir en action l'utilisation des
différents codes voir l'adresse: http://www.kazusa.
or.jp/ codon/
|
|||
|
Ce code est parfois dit ponctué car il possède un codon d'initiation (AUG) qui code pour la méthionine (en position interne) qui initie toutes les chaînes polypeptidiques (procaryotes et eucaryotes) et donc définit ce que l'on appelle le cadre de lecture (en effet, il suffit d'un décalage d'une base pour que les mots soient différents puisque le code n'est pas ponctué entre chaque mot); c'est la "phrase" que constitue l'information nécessaire à une séquence polypeptidique qui peut être éventuellement être qualifiée de ponctuée, puisqu'elle possède un signal d'initiation et un signal de terminaison (codons stop ou non-sens qui habituellement provoquent l'arrêt de l'allongement de la chaîne polypeptidique... mais l'on connaît des ARNt qui suppriment des mutations non-sens (voir par exemple certains antibiotiques utilisés dans le traitement de la mucoviscidose); de même le code est non chevauchant car les codons sont lus successivement sans chevauchement. Cependant il existe certains cas de chevauchement au niveau des gènes par décalage du cadre de lecture pour des ADN viraux. |
||||||
|
On remarque que les deux
premières bases de chaque codon sont pratiquement
déterminantes pour tous les ARNt, ce qui se
traduit par quatre codons possibles pour un aa typique. Plus
rarement deux codons sont associés à un aa et
l'on remarque cette fois que les deux bases puriques (A et
G) sont associés en 3ème position du codon
à un aa alors que les bases pyrimidiques (U et C)
sont associées de même à un autre aa.
Ces caractéristiques chimiques et structurales font
penser à des contraintes d'appariement entre les
trois bases de l'ARNm (codon dans le sens 5'->3') avec
les trois bases de l'ARNt (anticodon dans le sens inverse:
3'->5'), la première base de l'anticodon
s'associant avec la troisième base du codon. En fait
les ARNt possèdent un cinquième
nucléotide possible au niveau de leur anticodon :
l'inositate (renfermant une base inhabituelle:
l'hypoxanthine) qui peut s'associer (faiblement) par liaison
hydrogène avec U, C ou A. L'appariement le plus
lâche constaté entre la dernière base du
codon (de l'ADN) et de l'anti-codon (de l'ARNt) à
conduit F. Crick à émettre ce que l'on appelle
l'hypothèse du tremblement (wobble hypothesis)
qui énonce des règles d'associations
codon-anticodon et grâce à laquelle un nombre
minimal de 32 ARNt est nécessaire. On
considère que cet appariement lâche favorise la
vitesse de traduction sans diminuer la précision
étant donné la redondance du code.
|
||||||
|
Dans le tableau suivant les couleurs jaune - vert - bleu indiquent les degrés d'hydrophilie, notion expliquée dans la seconde partie (la logique du code génétique) |
L'échelle avec l'index d'hydropathie est une autre échelle d'hydrophilie (voir explications plus bas) |
|||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Index d'hydropathie* |
|||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
||
|
* échelle d'hydrophobie-philie EN SOLUTION d'après Kyte, J. et Doolittle, R.F. (1982), J. Mol. Biol., 157, 105-132 (in Principes de Biochimie, Lehninger, Nelson et Cox, 1994, Flammarion, 5-1) |
|||||||||||||||||||||
|
|
|
|
|||||||||||
|
|
|
|
|
||||||||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|||||
|
|
|
|
|
|
|
|
|
|
|
|
|
||
|
|
|
|
|
|
|
|
|
|
|
||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|||||
|
|
|
|
|
|
|
|
|
|
|
||||
|
|
|
|
|
|
|
|
|
|
|||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|||||
|
|
|
|
|
|
|
|
|
|
|
|
|||
|
|
|
méthionine |
|
|
|
|
|
|
|
||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|||||
|
|
|
|
|
|
|
|
|
|
|
||||
|
|
|
|
|
|
|
|
|
|
|||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
||||||
|
|
||||||
|
L'hydrophilie (affinité pour l'eau) / hydrophobie (répulsion pour l'eau) des aa ont été étudiées par Laurence Durst et Stephen Freeland (Pour la Science, Dossier n°46, janvier-mars 2005, p 15, figure 5). J'ai mis en regard en haut du tableau les valeurs données par une autre source (index d'hydropathie). |
Le code génétique contient une logique très forte si l'on considère que le changement d'une seule base (pour chaque codon) modifie modérément l'affinité pour l'eau de l'aa codé. Ainsi le changement de UCU (Ser) en ACU (Thr), CCU(Arg), GCU (Val) qui sont des aa fort différents a des conséquences relativement limitées du point de vue de l'affinité pour l'eau de ces aa (3 niveaux de l'échelle qui en comporte 8). |
Cette explication cadre très bien avec notre compréhension moderne de l'eau cellulaire (voir page sur la cellule) |
||||
|
|
||||||
|
Le code génétique, même s'il est exprimé en terme de correspondance ARNm-aa reflète en fait une liaison aa-ARNt. En effet, la correspondance entre un codon et un aa repose sur les ARNt et donc revient à établir une correspondance entre ARNt et les aa. On pense que la formation d'un complexe ARNt-aa résulte de l'activité d'enzymes (les ARNt synthétases) spécifiques de chaque couple (aa, ARNt). |
Il y a donc un ARNt spécifique pour chaque aa. On peut aussi différencier les ARNt par leur anti-codon, mais avec des redondances. On peut donc considérer qu'il y a deux domaines dans chaque ARNt, un domaine d'accrochage spécifique d'un aa - à cause de la présence d'une enzyme de type ARNt (l'enzyme est alors la cause de la correspondance aa-ARNt) - et un domaine de reconnaissance codon-anticodon (c'est la séquence de l'ARNt au voisinage du codon qui est spécifique de ce domaine). |
En supposant qu'il existe au départ un répertoire assez varié de domaine de reconnaissance ARNt-ARNm comment peut-on trouver un déterminisme d'accrochage de ces domaines avec le domaine de reconnaissance des aa ? |
||||
|
|
||||||
|
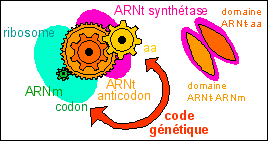
Une image pour montrer que le code génétique ne reflète pas une liaison directe entre ARNm et aa mais une liaison indirecte. |
Les
ribosomes
sont les structures à fonction
enzymatique qui relient l'ARNm aux complexes ARNt-aa et les
ARNt
synthétases sont les enzymes
spécifiques de aa et des ARNt.
|
 |
||||
|
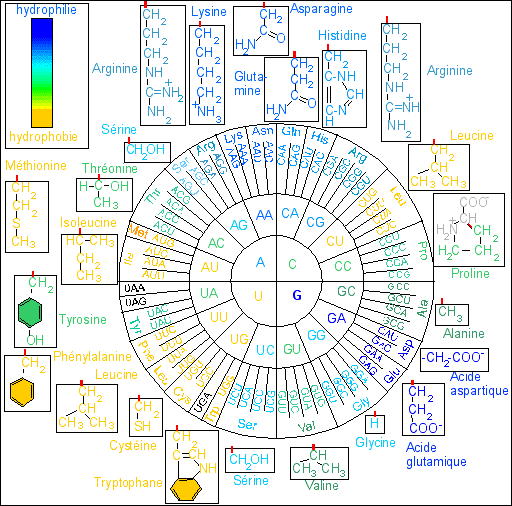
Une autre représentation du code génétique |
Le but de cette présentation n'est bien évidemment pas de trouver une relation entre séquence des codons et aa mais bien de regrouper les aa entre eux - comme la cellule le fait pour la synthèse des protéines - pour faciliter les recherches au niveau de l'ARNt.
|
 |
||||
|
+ les aa sont colorés en fonction de leur degré d'hydrophilie + les codons sont colorés de la même couleur que les aa qui leur correspondent - les codons STOP en noir + les aa ont pour formule H2N-CHR-COOH; seuls les groupements R, variables ont été représentés; sauf pour la Proline |
||||||
|
|
||||||