|
Remarques:
|
|
* Les enzymes sont classées en
fonction des réactions qu'elles catalysent.
Leur nom courant est souvent formé du nom du substrat
auquel on ajoute le suffixe -ase (exemple l'uréase
catalyse l'hydrolyse de l'urée). Mais chaque enzyme
possède un numéro de code (EC number) à
quatre chiffres (Classe / Sous-classe / Sous-sous-classe /
Ordre), un nom systématique et un nom commun
recommandé (http://www.chem.qmul.ac.uk/iubmb/enzyme/).
Les enzymes protéiques sont réparties en 6
classes: 1- les oxydoréductases, 2 - les
transférases, 3 - les hydrolases, 4 - les lyases, 5 - les
isomérases, 6 - les ligases.On notera que je n'ai pas
respecté l'orthographe ANGLOSAXONNE du radical "oxid" en
usage dans la classification internationale.
|
|
|
* La réaction enzymatique peut
être inhibée par la présence de
molécules qui l'inactivent ou diminuent sont
activité.
|
|
Un inhibiteur compétitif ne
modifie pas la vitesse de la réaction enzymatique en
présence de son substrat mais diminue l'affinité
apparente de l'enzyme avec son substrat lorsqu'il est
présent en solution. On interprète son action en
proposant que l'inhibiteur compétitif entre en
compétition avec l'enzyme en se fixant sur la partie de
reconnaissance du site actif sans pouvoir être
transformé. Selon son affinité il peut diminuer
plus ou moins l'activité enzymatique. Une comparaison
amusante que je tiens de mes années de classes
préparatoires: l'enzyme est comparée à un
mangeur de lentilles. Chaque cuillerée portée
à la bouche puis avalée est une réaction
enzymatique réalisée. Si l'on ajoute quelques
PETITS cailloux dans le plat de lentilles, le mangeur porte
à la bouche des cuillerées de lentilles avec une
belle régularité mais doit recracher certaines
cuillerées, comportant un petit caillou, ce qui diminue
grandement la vitesse de réaction, mais pas la vitesse
avec laquelle il porte les cuillerées à la
bouche... Le petit caillou joue le rôle d'inhibiteur
compétitif.
|
|
Un inhibiteur non compétitif ne
modifie pas l'affinité apparente de l'enzyme avec son
substrat mais uniquement la vitesse maximale de la
réaction. On interprète cette action comme le
résultat d'une fixation de l'inhibiteur non
compétitif sur un autre site que le site catalytique de
l'enzyme. Pour reprendre ma comparaison avec le mangeur de
lentilles, l'inhibiteur compétitif peut être
comparé à un GROS caillou dans les lentilles. Le
mangeur les voit et les évite, ce qui diminue
singulièrement sa vitesse de rotation des
cuillerées.
L'interprétation que l'on fait de l'action des
inhibiteurs va de pair avec celle des analogues de
structure qui sont des substances voisines (au sens
topologique) de molécules métaboliques ou
informationnelles habituelles mais qui bloquent ou diminuent
la fonction des molécules fonctionnelles ou
informatives auxquelles elles se lient. La mise au point
d'analogues de substrat est une des étapes essentielles
de la recherche pharmaceutique.
|
|
TP
classique :
hydrolyse de l'ovalbumine par une pepsine
|
|
En plus de la manip faite en TP on peut
améliorer la compréhension du modèle
moléculaire de l'activité enzymatique grâce
à des outils de modélisation :
- on peut télécharger le fichier .pdb de
l'ovalbumine (1ova.pdb) sur le site http://www.rcsb.org/pdb/
- à l'aide de ce fichier, il est possible de rechercher
les liaisons hydrolysées grâce à l'enzyme :
il s'agit des liaisons adjacentes à la tyrosine ou
à la phénylalanine. En effet, la pepsine
catalyse préférentiellement la rupture des
liaisons peptidiques où un acide aminé
aromatique est engagé par son groupement aminé.
Je précise que la pepsine est une enzyme naturelle
extraite du pancréas de porc (aussi
nommée gastérase...http://www.biam2.org/www/Sub2922.html)
.
Sa dénomination internationale est pepsin
E.C.3.4.23.1 (un fichier .PDB complet d'une pepsine est
par exemple 4pep.pdb (1990) (http://www.rcsb.org/pdb/explore/explore.do?structureId=4PEP),
mais je recommande aussi le fichier Kinemage: 4pep.kin
(voir la page http://www.rcsb.org/pdb/explore.do?structureId=4PEP
et http://www.rcsb.org/pdb/files/4PEP.kin
pour télécharger directement le fichier (à
enregistrer au format "text" avec l'extension ".kin" et à
ouvrir dans l'application KING; pour télécharger
l'application java, voir les indications sur la page http://kinemage.biochem.duke.edu/software/index.php
)
|
|
|
|
|
3 - La
fonction enzymatique est une "fonction topologique"
|
|
|
|
|
Je rappelle que le mot fonction
a tout intérêt à être
défini au sens mathématique de la façon
la plus rigoureuse qui soit. Une fonction biologique c'est
un phénomène qui peut être
représenté par une fonction
mathématique (voir cours
de seconde et une
petite page sur les mathématiques en SVT en seconde).
Ici nous nous intéressons à des fonction
locales et donc que l'on peut explorer
expérimentalement.
|
|
La topologie est une
branche des mathématiques qui s'intéresse aux
propriétés de l'espace et des ensembles de
définition des fonctions du seul point de vue
qualitatif; elle précise les notions de
continuité-discontinuité, bords, limites...
|
|
|
3.1 Comprendre la fonction enzymatique
|
|
|
|
|
|
|
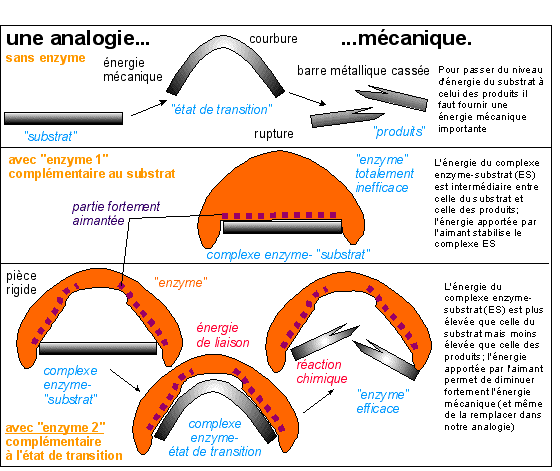
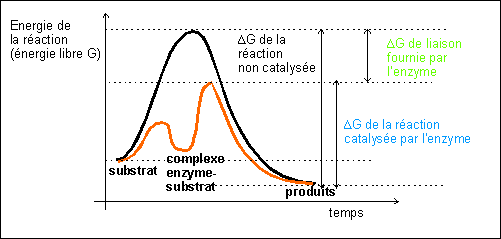
Être une enzyme signifie avoir une fonction
enzymatique. C'est-à-dire une fonction
catalytique (accélération
d'une réaction chimique sans altération du
catalyseur). On pourrait dire que cette
fonction est topologique dans le sens où
elle repose sur une similitude de forme entre l'enzyme et un
état transitionnel du(des) substrat(s).
|
|
|
Mais
comment comprendre l'activité enzymatique ? Une fois
encore on a (au moins) deux attitudes (voir
aussi la page de synthèse sur les
niveaux d'organisation du vivant)
:
|
|
- chercher à comprendre comment est "contrôlée"
la forme de l'enzyme (et plus particulièrement du site
actif) et en quoi cette forme permet l'activité
catalytique au niveau chimique (liaisons faibles, transfert de
charges...). C'est la voie du réductionnisme.
Je vous propose une autre voie.
|
|
- accepter comme un fait
expérimental (empirique) ce contrôle de la
forme et chercher à en formaliser les limites. C'est la
voie du structuralisme. Toute fonction topologique -
qui repose sur une correspondance des formes - pourrait
être appelée enzymatique. René Thom en a
dessiné les contours (dans Modèles
mathématiques de la morphogénèse (1971,
1974, 1980) notamment).
|
|
|
|
|
|
|
Voici quelques exemples de ces
deux attitudes:
|
|
voie réductionniste
(c'est la voie de
vos manuels scolaires et de l'immense majorité des
recherches):
|
|
La correspondance entre les formes du site
actif de l'enzyme et de la forme de transition du substrat
conduit à l'établissement de très
nombreuses liaisons faibles (Van der Wals, hydrophobe...) qui
sont responsables de l'apport d'une énergie qui
expliquerait l'activité enzymatique elle-même.
Cette manière de voir, encore bien mystérieuse,
est plus qualitative que quantitative. Pour essayer de
réduire l'activité enzymatique à des
phénomènes moléculaires connus on peut
considérer qu'elle repose sur la capacité de
molécules à établir des liaisons ou
à les rompre, ce qui est la base de toutes les
réactions chimiques. Cette capacité
dépend bien sûr des propriétés
propres de la molécule (répartition de ses
charges dues à son composants et à ses groupes
caractéristiques (que l'on appelait auparavant
improprement "fonctions"), ... ) mais aussi des conditions
physiques du milieu (pression, température,
ligands, charges voisines dont surtout molécules
d'eau...).
|
|
L'activité enzymatique dépend
donc d'une part de conditions propres
de la molécules (internes ou
intrinsèques) et de conditions du
milieu (externes ou
extrinsèques). Il y a un réductionnisme
interne et externe (voir
René Thom, Modèles mathématiques de la
morphogénèse, p 42 et s).
Pour une protéine enzymatique on relie habituellement
les conditions propres à plusieurs niveaux
d'organisation:
- la structure primaire qui est la
séquence des aa de chaque chaîne de la
protéine (qui peut en comporter plusieurs): elle est
due aux liaisons COVALENTES : liaisons peptidiques (-CO-NH-)
entre les groupes fonctionnels carboxyle (-COOH) et amine (-
NH2) des aa et liaisons disulfure (-S-S-
) entre les radicaux (-SH) des aa cystéine (Cys).
- les structures d'ordre supérieur (2 à
4) qui correspond au repliement de chaque chaîne et
à leur association entre elles (et
avec des groupements supplémentaires dits prosthétiques
pour les hétéroprotéines)
par des liaisons FAIBLES.
La structure primaire est en relation indirecte (par
l'intermédiaire du code génétique, des
ribosomes, des ARNt) avec l'information
génétique des ARNm recopiée
à partir de celle de l'ADN. On sait modifier - par
génie génétique - l'information
génétique ou des étapes de la
synthèse des protéines au sens large (de la
transcription, de la maturation des ARNm ou de la traduction)
afin de faire produire à une cellule des enzymes
à activité réduite ou supprimée.
|
|
MAIS,
plus on avance dans la connaissance des protéines moins
il devient évident de relier la structure primaire avec
la forme de molécule; au contraire, les formes des
protéines sont caractérisées par un nombre
limité de formes de base
(ou motifs - voir par
exemple Les protéines, rouages des cellules, H.
Berman, D. Goodsell et P. Bourne, Pour la Science,
Dossier n°46, janvier mars 2005, p 32;
plus récemment voir Sebastian E. Ahnert, Joseph A.
Marsh, Helena Hernandez, Carol V. Robinson, Sarah A. Teichmann
Principles of assembly reveal a periodic table of
protein complexes
Science 350, 1331 and aaa2245 (2015). DOI:
10.1126/science.aaa2245 : 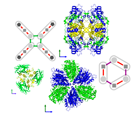
http://www.periodicproteincomplexes.org )
qui sont obtenus pour des séquences très
différentes étant donné les profondes
similitudes des aa vis-à-vis du composant majoritaire
de la cellule: l'eau (voir
remarque ci-dessous et la page code
génétique).
Avec la démarche inverse il est clair que la
prédiction (complète) de la structure d'ordre
supérieur à partir de la structure primaire est
impossible. Par contre, si l'on s'appuie sur une
théorie du type de celle de la théorie des
catastrophes, il devient possible de prévoir la forme
d'une molécule à partir de sa fonction (et non
plus de sa séquence primaire), comme on peut le faire
pour une protéine enzymatique (voir paragraphe
ci-dessous: voie structurale).
Il est aussi important de ne pas confondre la stabilité
de la forme (ou conformation stable au sens de forme
dans l'espace) d'une protéine isolée (ce
que l'on peut relier à sa capacité à
cristalliser - c'est-à-dire former un cristal
ordonné -, et qui dépend bien sûr de sa
structure aux différents niveaux)
et l'ordre (voir Le
désordre, clé de voûte des
protéines, Véronique
Receveur-Béchot et David Karlin, La Recherche, juin
2005, 387, 52-55). De nombreuses
protéines (probablement plus
de la moitié des protéines connues)
présentent pour la totalité, ou pour une partie
de leur séquence, des zones qui ne se replient pas
(appelées maladroitement "désordonnées"
ou pire "non structurées" ) et qui n'en sont
pas pour autant moins essentielles dans la fonction de la
protéine. Les fonctions associées sont encore
insuffisamment vulgarisées mais il est probable que
l'on découvre une grande diversité de fonctions
mécaniques (l'article de La
Recherche cite notamment la titine, protéine
géante, assurant la cohésion des
myofibrilles...) ou chimiques. Ces
protéines n'auraient pas une activité de type
enzymatique au sens défini ci-dessus de "fonction
topologique" mais bien des fonctions mécaniques,
électriques ou chimiques variées: ce ne seraient
pas des enzymes mais des protéines
fonctionnelles (ayant une fonction propre
liée à leur structure mais pas à une
structure trimensionnelle ou conformation spatiale STABLE).
Les conditions externes de l'activité enzymatique sont
très mal connues IN VIVO.
|
|
|
|
|
|
|
Remarque:
L'élément essentiel dans une cellule vivante est
l'eau (une cellule comprend 80% d'eau environ). Mais cette eau
n'est pas libre et n'est donc pas disponible comme solvant.
Les modèles d'activité enzymatique
calculés à partir d'expériences in
vitro, en solution aqueuse sont inappropriés pour
le vivant (et plus encore les logiciels de simulation -
strictement "abio"chimiques - qui ne représentent
jamais les complexes enzymatiques pourtant omniprésents
dans les cellules).
On sait qu'une couche d'eau extrêmement fine (d'une
épaisseur de quelques molécules soit 1,1 nm
environ - voir page
sur la cellule) entoure toutes
les macromolécules cytoplasmiques. On l'appelle "eau
interfaciale". Cette eau peut favoriser ou au contraire
défavoriser la liaison enzyme-substrat. On peut aller
jusqu'à dire (en restant dans une approche
réductionniste) que c'est cette eau interfaciale
qui contrôle l'activité enzymatique.
|
|
|
|
voie structurale
(je
précise que cette voie est peu balisée
- et beaucoup auront du mal à différencier
réductionnisme externe et structuralisme -
mais ô combien attirante):
|
|
|
Si la fonction enzymatique est topologique (au
niveau moléculaire) on va donc pouvoir trouver - pour
une enzyme donnée - une autre molécule,
qui ne soit pas forcément une protéine (et qui
habituellement n'a pas d'activité chimique
réactionnelle) mais qui va pouvoir présenter
une fonction catalytique due uniquement à
sa forme.
|
|
On a d'abord découvert des ARN à
fonction enzymatique (les ribozymes) qui peuvent
catalyser la coupure d'autres ARN
(ARNase P) ou même ce que l'on appelle improprement une
autocatalyse (les molécules d'ARN
"autocatalytiques" de certains virus ou fabriquées
artificiellement sur ce modèle étant
susceptibles de se couper elles-mêmes en plusieurs
fragments) - un catalyseur par définition doit
être restitué intégralement en fin de
réaction ce qui n'est pas le cas dans une
"autocatalyse". Mais la connaissance
topologique de ces "enzymes" reste insuffisante.
|
|
C'est Linus Pauling en 1940 qui émit
une hypothèse beaucoup plus générale et
qui va tout à fait dans le sens structural: il
suggéra de rechercher des anticorps (notés
Ac; ce sont des glycoprotéines formées de 4
chaînes associées en forme d'"y" - voir
immunologie en classe de terminale) dirigés
contre l'état de transition d'un substrat
réactionnel, qui devraient donc être
doués d'une activité catalytique. On a
leur a donné le nom d'abzymes. Vous
remarquerez aisément que l'on n'a aucun moyen d'isoler
le site actif d'une enzyme. On est donc bien obligé de
passer par des l'état de transition. Mais comme ce
dernier est beaucoup trop instable pour être
observé il faut bien passer par des analogues de
l'état de transition. On les repère en cherchant
des molécules qui se fixent au site actif d'une enzyme
avec des affinités encore beaucoup plus importantes que
le substrat réactionnel et qui y restent...(ce sont des
inhibiteurs de type bloquants : ils réalisent une
inhibition irréversible). Cependant, à partir de
1974, lorsque la notion de réseau idiotypique a
été formalisée par Niels Kaj, on a
élargi la recherche des analogues à celle des Ac
dirigés contre des épitopes des Ac
dirigés contre une enzyme et ainsi produire des Ac
dirigés spécifiquement contre le site actif de
l'enzyme. Il a fallu attendre 1986
pour obtenir les premières abzymes. Les
molécules d'Ac ont naturellement une diversité
topologique quasi infinie. On produit notamment des abzymes en
sélectionnant (de façon
indirecte par des molécules qui fixent elles aussi ces
mêmes Ac) des Ac dirigés
vers le site actif d'enzymes (sorte de moule interne du site
actif). Puis on fabrique des Ac complémentaires de ces
Ac (moule externe de ces moules internes) et qui
possèdent alors une activité catalytique voisine
de l'enzyme.
|
|
|
On peut élargir le
concept d'enzyme à toute structure
qui, par correspondance topologique avec un état
transitionnel, permettrait une activité.
C'est ce que Thom appelle l'effet enzymatique, si je ne
me trompe.
|
|
Deux extraits de Stabilité
structurelle et morphogénèse - Essai d'une
théorie générale des modèles,
1968 (stabilite.pdf)
|
|
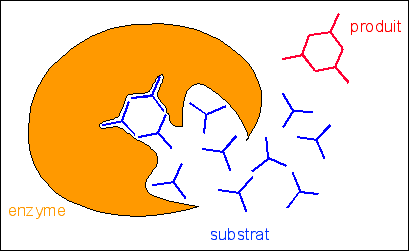
« Dans un autre exemple tiré
des solides, le système clef-serrure cher aux
biochimistes, il ne peut y avoir interaction qu'à
condition de supposer la clef mobile (dans toutes les
positions permises dans le complémentaire de la
serrure) ; une clef n'est porteuse de signification pour une
serrure que si l'on l'enfile et on la tourne. On a là
un exemple typique d'une situation où, si l'on prend
clef et serrure statiquement, presque toute la structure de
leur interaction repose sur l'agent moteur, l'individu qui
tourne la clef, autrement dit sur le potentiel d'interaction
; si l'on veut se placer dans une optique qui élimine
l'interaction, il faut rendre les deux systèmes
mobiles ». (p 187)
|
|
« B. Morphologie et Biochimie
On peut d'ailleurs se demander d'ores et déjà
si, au lieu d'expliquer la Morphogénèse par la
Biochimie, ce n'est pas l'attitude inverse qu'il convient de
prendre. Le problème de déterminer la structure
tertiaire d'une protéine en fonction de sa structure
primaire est, typiquement, un problème de dynamique des
formes (au sens du chapitre 7) ; il s'agit de
déterminer les minima de la fonction U, énergie
libre, sur l'espace fonctionnel L de toutes les configurations
de la molécule ; qui pourrait douter que l'ensemble S
de bifurcation de L, qui représente l'ensemble des
positions où la molécule se recoupe, ne joue un
rôle de singularité pour U? Et que la topologie
relative des bassins des différents minima de U ne joue
un rôle essentiel dans l'activité, enzymatique ou
motrice, de la molécule ? L'étude récente
de diverses enzymes (le lysozyme, par exemple) a montré
l'aspect éminemment morphologique de bien des
réactions enzymatiques ; on voit les
molécules se palper, se pincer, se tordre, se
déchirer presque comme des êtres vivants ;
il ne faut pas s'en étonner ; dans la mesure où
une réaction biochimique reflète un incident
local d'une compétition spatiale entre
différents régimes, les contraintes
topologiques imposeront à ces incidents locaux de simuler
les catastrophes globales de la morphogenèse sur
l'espace-temps R4. En bien des cas, ces
mécanismes moléculaires se sont imposés
parce qu'ils réalisaient la meilleure simulation locale
de la catastrophe biologique globale qui leur a donné
naissance. Le fait que certains de ces mécanismes persistent
in vitro ne doit pas dissimuler leur origine et
leur signification biologiques. Ces réactions
présentent alors une grande stabilité
interne vis-à-vis du milieu ambiant ; elles
perdent d'ailleurs in vitro l'aspect polarisé et
orienté qu'elles présentent en milieu vivant. Il
est frappant d'observer à cet égard, que les
réactions de réplication génétique
affectant l'ADN - dont on a voulu faire l'essence même
de la vie - sont relativement grossières et
résistantes, alors que les réactions qui
dominent toute l'énergétique vitale, comme
celles qui ont lieu dans les plastides et les mitochondries,
dépendent d'une morphologie qu'on n'a pu jusqu'à
présent reproduire in vitro.» (p 216)
|
|
Il n'est pas faux de dire que la tentative
pour comprendre les enzymes "à la manière de
Thom" s'apparente à une redécouverte de la
notion d'enzyme-propriété qui s'est
longtemps opposée à celle d'enzyme-substance
(cf EU article "enzymes"); l'histoire est un éternel
recommencement. Le projet est bien d'unir en dépassant
l'apparente séparation entre ces deux notions.
Si l'on reste au niveau local on ne voit que des
discontinuités (liaisons établies ou rompues)
alors qu'au niveau global, analogique du niveau du vivant, on
voit des enzymes reproductrices (qui réalisent
des isomorphismes... comme une ADNpolymérase (ou
plutôt tout le complexe réplicatif dont l'ADNpol)
qui fabrique une copie d'une molécule... ), des enzymes
relationnelles (protectrices (chaperonines... par
isolement de la structure à protéger),
informatrices (par déformation d'une cible et non pas
reproduction d'une déformation...)....) et des enzymes
nourricières (qui transfèrent de la
matière ou de l'énergie...)...
|
|
|
3.2 L'exemple des polysomes et essai de
généralisation
|
|
|
|
pour
les données moléculaires simples de niveau
lycée voir la page :
le
gène moléculaire, unité fonctionnelle
synthétique
|
|
Un exemple particulièrement simple
à comprendre est celui des ribosomes. Ces
particules sont maintenant très bien connues et l'on peut
affirmer que d'une part ce sont bien leur
propriétés topologiques qui leur
confèrent leur fonction et d'autre part que ces
complexes ARN-protéines sont restitués
intégralement sans altération en fin de
réaction (ce que l'on appelle le cycle ribosomial):
ces deux propriétés en font des enzymes au sens
général. À ce sujet on dit souvent que les
ribosomes sont les catalyseurs de la synthèse
protéique, ce qui correspond bien à les apparenter
à des enzymes.
À la limite on pourrait aussi considérer que
la propriété de l'ARNm qui est de servir de
modèle pour l'enchaînement des aa est une
propriété de type enzymatique car topologique et
non destructrice. L'information génétique
serait alors topologique ou encore enzymatique. La fonction
globale est bien ici une reproduction.
Seul
l'espace des phases permet d'approcher les dynamiques.
|
|
 Les
éléments qui suivent sont repris de la conférence
du 4e congrès BCM - Poitiers 2005
(voir la 2ème partie)- la réflexion y est plus
complète Les
éléments qui suivent sont repris de la conférence
du 4e congrès BCM - Poitiers 2005
(voir la 2ème partie)- la réflexion y est plus
complète
|
|
On est tenté d'assimiler le plan (x,y)
des observations au plan (x,t) des positions, mais cela n'est
pas possible car on a un cadavre fixé et coloré.
Les physiciens utilisent l'espace
des phases* (voir
page
sur le continu).
Pour un point animé d'un mouvement dans un plan ,
l'espace des phases est un plan (x,v). On pourrait alors avoir
une spirale divergente (ou convergente) dans le cas d'un
système linéaire amorti. Dans l'espace des
positions on a une trajectoire linéaire bornée
aux deux extrémités, ce qui n'a rien à
voir avec notre "spirale". Pour obtenir une telle morphologie
la dynamique est donc plus compliquée (deux
attracteurs** au moins doivent s'affronter). Il faut
approfondir ses bases mathématiques et surtout
géométriques et analytiques.
*espace abstrait qui permet de définir
complètement l'état d'un système à
partir de la connaissance des coordonnées de ce
système dans cet espace
** attracteur; régime asymptotique
stable (atteint au bout d'un temps infini)
|
|
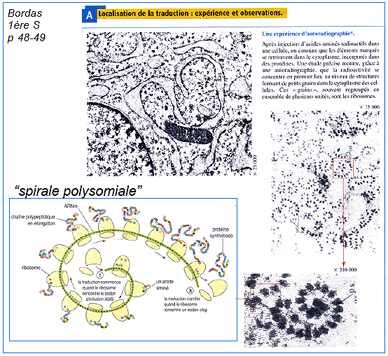
|
|
|
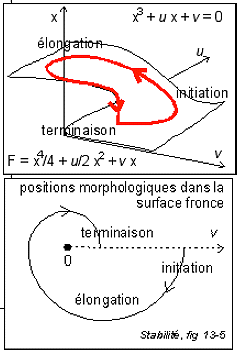
La fronce permet une
représentation d'une dynamique analogue à la
dynamique polysomiale dans un espace des phases dont les
paramètres sont à préciser
|
|
|
On notera que la dynamique proposée est
récurrente pour les modèles thomiens
(peut-être parce qu'elle représente un premier
niveau d'approche - qu'il est bien sûr nécessaire
de la préciser, ce qui ne peut être fait à
ce niveau de compréhension - mon propos ici n'est que
de souligner l'analogie en me servant d'un outil... que je ne
maîtrise pas mais dont je vois l'utilisation que
pourrait en faire un mathématicien). L'arbre
à came de droite est analogue à la forme
des polysomes dans l'espace
cytoplasmique fixé et mort.
|
On peut penser que dans ce cas la dynamique
proposée est pertinente et qu'il ne reste qu'à
trouver les paramètres qui ont été
figés dans la coupe. Si le système de
synthèse protéique est stable il n'est pas
étonnant que la surface fronce représente une
coupe de l'espace euclidien. Il est urgent de s'efforcer de
trouver des paramètres pertinents de la dynamique
métabolique cytoplasmique. C'est un travail que je ne
peux faire seul. J'en appelle une fois encore à
l'inventivité de notre jeune génération.
On remarquera que l'initiation est habituellement
représentée au centre de la figure mais je ne
connais aucun argument en faveur de cette hypothèse. La
dynamique présentée ici exige que l'initiation
se fasse sur le bord externe du polysome
(l'extrémité 5' de l'ARNm avec sa coiffe
("cap structure") doit se trouver à cet endroit et non
au centre). Je souligne deux points. D'une part, une dynamique
convergente (même si ce n'est pas dans l'espace
euclidien) est plus parlante et conforme à l'intuition
(la libération des sous-unités ribosomiales au
point de potentiel le plus bas permet aisément une
récupération au niveau du site d'initiation
voisin). D'autre part, on peut penser qu'il existe une
fonction de structuration de la protéine au sein du
polysome, les ribosomes pouvant intervenir comme "chaperons".
Ceci est une piste de recherche ouverte (interactions
sous-unités ribosomiales - polypetide en formation). En
tout cas la localisation du point d'initiation devrait pouvoir
être assez facilement précisée
expérimentalement.
|
|
La fronce
Une représentation de la morphologie
de la dynamique du cycle ribosomial (la surface est le lieu
des points singuliers du potentiel F, c'est-à-dire le
lieu où la dérivée s'annule, voir
page
complémentaire pour des
explications simples avec des exemples.)
|