2. Cellules et molécules du
vivant
retour plan du cours de
1èreS
Plan de cette page:
- 2.1 Le gène moléculaire,
unité fonctionnelle cellulaire
- a. L'information génétique stable est
contenue dans l'ADN et est copiée dans l'ARN lors de
la transcription
- b. L'information génétique transitoire des
ARNm est traduite en polypeptides (à l'aide
d'autres ARN) dans les ribosomes du cytoplasme
- c. Les peptides exportés subissent une maturation
dans le REL puis dans l'appareil de Golgi avant d'atteindre la
membrane ou d'être sécrétés par
exocytose
- d. L'information génétique est une
information linéaire pour une molécule
- 2.2 Un exemple de molécules
fonctionnelles: les protéines enzymatiques
- 2.2.1 Les enzymes sont des biocatalyseurs: elles
catalysent des réactions biochimiques (du vivant) qui
appartiennent au métabolisme
- 2.2.2 La réaction enzymatique se réalise
à l'intérieur d'une poche appelée site
actif.
- 2.2.3 La fonction enzymatique est une "fonction
topologique"
- 2.3 Du phénotype au
génotype et retour
- 2.3.1 Un peu d'histoire: : l'embrouille
héréditaro-moléculaire
- 2.3.2 Des espoirs déçus aux espoirs
naissants
- 2.3.3. Retour à la cellule et au travail du
vivant
Compléments:
* page annexe Phénotypes et
génotypes
* page annexe: les 4 causes d'Aristote en
sciences de la vie
2.1. Le gène moléculaire,
unité fonctionnelle cellulaire
La notion de gène moléculaire a
été vue en classe de seconde (le
gène moléculaire, au sens de la biologie
moléculaire, n'est pas l'équivalent du gène
héréditaire, portion de chromosome associée
à un caractère héréditaire - voir
cours de
seconde). Il s'agit ici de la
développer.
Remarque de vocabulaire: les chaînes d'aa sont des
peptides (aa réunis par des liaisons peptidiques). Les
oligopeptides sont formés quelques aa (10...); les
polypeptides sont formés de nombreux aa (10-100) mais
lorsque leur poids moléculaire est supérieur à
10.000 on préfère parler de protéines.
Les protéines sont souvent formées de l'assemblage de
plusieurs molécules et contiennent des éléments
non peptidiques (appelés groupement
prosthétique, comme le groupe tétrapyrolique
à Fe qui forme l'hème et s'ajoute à la
molécule de globine de l'hémoglobine... elle-même
formée de 4 sous-unités identiques 2 à
2).
TP-TD - Acides aminés et
protéines; simualisation* en 3D
(attention applet Jmol de 540Ko à
télécharger)
(* les superbes
applications qui visualisent les molécules en 3D sont des
simulations; pour souligner ce fait je parlerai de
simualisation)
a. L'information génétique stable est contenue dans
l'ADN et est copiée dans l'ARN lors de la transcription
* Les ARN sont des acides nucléiques
Les acides nucléiques sont l'ADN et les ARN
(voir cours
de seconde).
L'ADN est une double chaîne de
désoxyribonucléotides enroulée en
hélice.
Les ARN (acides ribonucléiques) sont des
chaînes de ribo-nucléotides. Elles n'ont donc
qu'un seul brin (ce sont des chaînes simples ou monobrin). Les
nucléotides de l'ARN, par comparaison à ceux de l'ADN,
comportent de l'Uracile (U) à la place de la Thymine (T) et du
ribose à la place du désoxyribose.
Comme pour les composants de l'ADN, un
ribo-nucléotide est association d'une base
azotée (A=adénine, C=cytosine, G=guanine, U=uracile),
d'un sucre (le ribose) et d'un, deux ou trois groupement phosphate
(PO43-).
Comme pour les composants de l'ADN, un ribo-nucléoside
est composé d'une base (A,U,C ou G) et du sucre(le
ribose).
Pour simualiser les acides nucléiques et leurs composants voir
le TP e
seconde.
Les nucléotides ne sont pas que des composants des acides
nucléiques mais ont des fonctions variées dans la
cellule: par exemple l'ATP (adénosine triphosphate =
adénine + ribose (l'adénosine est le nom du
nucléoside) + 3 phosphates) et ses dérivés
monophosphate (AMP) ou diphosphate (ADP) qui sont des
molécules énergétiques intermédiaires
(transfert de l'énergie chimique de liaison entre
molécules) mais aussi informatives (AMP cyclique ou cAMP par
exemple); ou le dGTP (adénine+désoxyribose+3
groupements phosphate; le "d" devant la
molécule indiquant la nature du sucre: le
désoxyribose) qui est aussi une molécule
énergétique.
* L'ADN nucléaire est transcrit en ARN par des complexes
enzymatiques (ARN polymérases) au sein du noyau
La transcription est la copie de l'un des brins de l'ADN en
ARN. C'est donc la synthèse d'une molécule d'ARN
(monobrin) à partir d'un l'un des brins d'une molécule
d'ADN (brin matrice servant à une copie
complémentaire base à base).
Lors de la transcription, la molécule d'ADN est
maintenue ouverte (brins séparés) par un gros complexe
enzymatique : l'ARN polymérase (ARN polymérase
ADN dépendante qui contient du Zn2+) dans une zone d'une
longueur de 17 paires de bases (3,4 nm pour 10 pb) ce qui
nécessite, du fait de l'enroulement de l'ADN, de
dérouler l'ADN en amont et de le réenrouler en aval de
la zone de transcription. L'ARN polymérase copie un des brins
de l'ADN (brin non codant ou brin ADN matrice ou brin transcrit) en
un ARN complémentaire à une vitesse qui atteint 50
nucléotides à la seconde chez E. coli. La transcription
nécessite l'ion Mg2+ et de l'énergie fournie par les
ribonucléosides triphosphate (ATP, GTP, CTP et UTP).
Toutes les étapes de la transcription sont
régulées par des enzymes ou des facteurs par de
nombreux mécanismes qui constituent le cœur de la
régulation de l'expression génétique.
Les gènes moléculaires
exprimés sont transcrits et les
gènes moléculaires
réprimés ne sont pas
transcrits.
L'expression de l'information génétique
résulte directement de l'activité de
transcription.
Contrôler l'expression de l'information
génétique c'est d'abord contrôler la
transcription.
|
Il existe plusieurs types d'ARN. Pour simplifier on ne parlera que
des ARN messagers et des "autres ARN" pour désigner les ARN
qui ne sont pas traduits.
Si l'ensemble des gènes moléculaires constitue le
génome, les gènes
moléculaires qui sont transcrits en ARNm (qui seront traduits
à leur tour en protéines) constituent le
protéome.
Remarque:
Une molécule d'ADN peut être transcrite
simultanément par plusieurs molécules d'ARN
(voir par exemple Bordas doc 4 p45 ou Nathan
doc2a p 52).
* Les ARN messagers (ARNm) migrent du nucléoplasme vers
le cytoplasme
Les ARN messagers ou ARNm, migrent rapidement dans le cytoplasme
par les pores nucléaire.
Remarques:
1 - L'ADN des mitochondries et des chloroplastes est transcrit
directement au sein de ces organites.
2 - Les ARNm subissent de nombreuses maturations avant d'être
éventuellement traduits.
3 - L'ensemble des ARNm d'une cellule forme le transcriptome;
pour un fibroblaste humain par exemple il peut comprendre
jusqu'à 300.000 molécules d'ARNm (une
cellule humaine comprenant 50.000 gènes moléculaires
environ mais on pense que seuls 10.000 à 20.000 d'entre eux
sont actifs dans une cellule différenciée),
chaque ARNm n'étant présent que sous la forme d'au
maximum 15 copies. On sait détecter par la technologie des
puces à ADN (DNA chips)
une unique copie d'un ARNm dans un broyât cellulaire...
4 - L'information génétique contenue dans l'ARN peut
passer dans l'ADN par transcription inverse grâce
à des molécules de type ADNpolymérase
ARNdépendante (ou transcriptase inverse): voir le cours
d'immunologie de TS sur le VIH. Depuis les années 80-90
où on les recherche, on a trouvé des transcriptases
inverses chez des bactéries, des mycètes et même
chez les ovocytes de poissons.
5 - L'ADN peut être copié (répliqué) par
des enzymes (ADN polymérase) (cours de seconde). L'ARN peut
aussi être répliqué par des ARN
polymérases. Au total on a donc 4 types de synthèses
d'acides nucléiques: 2 réplications,
isosynthèses à partir d'un brin de même nature
que le brin synthétisé, et 2
hétérosynthèses à partir d'un brin
matrice de nature différente.
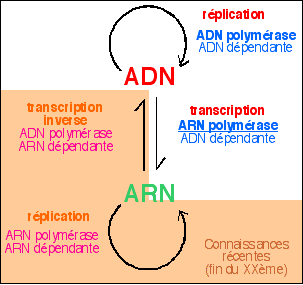
On notera cependant que les complexes enzymatiques
ARN dépendants (en rose) n'ont pas été
trouvés dans toutes les cellules et que ces voies ne sont pas
généralisables. Le fait qu'elles existent ouvre la voie
à une nouvelle compréhension de la notion d'information
génétique.
Les découvertes datant de
la fin du XXème siècle remettent en cause le dogme
central de la biologie moléculaire d'alors selon lequel
l'information génétique est contenue dans le seul ADN
est exprimée dans l'ARN. Si des ARN peuvent modifier de
façon importante l'ADN (ce qui n'est peut-être pas le
cas, car, pour l'instant, ce sont uniquement des ARN courts que l'on
sait s'insérer dans le génome), l'information
génétique doit donc comprendre à la fois l'ADN
et l'ARN; voir la page
sur l'histoire de la
génétique
pour les terminales S). Cependant la molécule d'ADN semble
être plus stable que les molécules d'ARN. Enfin, on peut
étendre aux protéines la notion d'information
génétique si l'on se réfère au
protéome (voir
cours de seconde). L'ADN a
été longtemps consifdérée comme la seule
infortmation génétique à caractère
héréditaire étant donné qu'elle
était la seule stable. Mais si l'ARN, qui est aussi transmis
héréditairement, peut transmettre une information
génétique stable par transcription inverse, cela change
fondamentalement les données du problème de
l'hérédité moléculaire.
b. L'information génétique transitoire des ARNm
est traduite en polypeptides (à l'aide d'autres ARN) dans les
ribosomes du cytoplasme
Les ribosomes sont de petits (20 nm environ,
un peu plus petits chez les procaryotes, un peu plus
gros chez les eucaryotes) complexes ARN-protéines
composés de 2 sous-unités qui s'assemblent lors de la
traduction.
Les ribosomes sont associés à un ARNm en
polysomes. Un polysome se présente au MET comme une
chaîne (parfois fixés au MET sous la
forme d'une spirale) de ribosomes le long d'une
molécule d'ARNm qu'ils traduisent.
Les polysomes sont soit libres dans le cytoplasme, soit
associés à la surface du RE sacculaire qu'on appelle
alors REG (reticulum endoplasmique granuleux ou granaire).
Lors de la traduction les aa sont présentés
aux ribosomes sous une forme activée (qui nécessite de
l'ATP) et associés à des ARN transporteurs-adaptateurs
(de transfert = ARNt). Ils sont liés progressivement les uns
aux autres par des liaisons covalentes (peptidiques) dans l'ordre
déterminé par l'ARNm. En effet, chaque triplet de base
de l'ARNm (codon) est associé à un aa activé
grâce à son ARNt transporteur-adaptateur
spécifique. La correspondance entre les codons de l'ARNm et
les aa constitue le code
génétique.
Le code génétique est universel (toutes les
systèmes de traduction cellulaire utilisent les mêmes
correspondances codon-aa). Cette universalité n'est pas
absolue mais la rareté des exceptions (par exemple dans les
mitochondries) prouve au contraire que ces exceptions sont
plutôt des optimisations que des dérogations à la
règle (il n'y a qu'une dizaine d'aa dans les
peptides traduits dans la matrice mitochondriale et les ARNt
mitochondriaux sont issus du génome mitochondrial)
prouve au contraire l'importance de l'universalité du code:
tout changement a des conséquences très graves pour le
sens de l'information génétique de la cellule.
Le code génétique est parfois dit
dégénéré car notamment
redondant (plusieurs codons correspondent à un aa)
(mais ce terme se réfère à une étape
antérieure qui n'a probablement pas existé) et
ponctué (codon initiateur et codon stop; mais ce terme
se réfère à l'expression du message et non au
code lui-même).
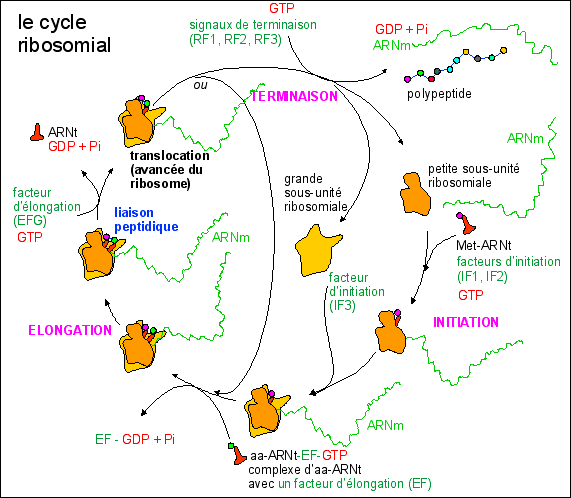
Les ribosomes lient les aa correspondant à deux codons
successifs puis progressent le long de l'ARNm en se décalant
d'un codon. La traduction comporte un phase d'initiation
(codon initiateur AUG correspondant à la Met), une phase
d'élongation (20 aa par seconde environ), et une phase
de terminaison (codon stop: UGA, UAA, UAG). La traduction est
un mécanisme métabolique qui consomme de
l'énergie (GTP) et qui est contrôlé par de
nombreuses enzymes.
Les ARNm sont traduits plusieurs fois avant d'être
détruits.
La plupart des protéines sont organisées en
complexes fonctionnels qui s'auto-assemblent sous le contrôle
d'enzymes et de facteurs spécifiques (par exemple les
molécules chaperons qui assurent le repliement de certaines
protéines...).
Remarque: Les mitochondries et les
chloroplastes possèdent des ribosomes qui réalisent la
traduction des ARNm directement dans leur compartiment matriciel. De
nombreuses protéines des ces compartiments possèdent
des sous-unités codées à la fois par le
génome nucléaire et le génome mitochondrial ou
chloroplastique.
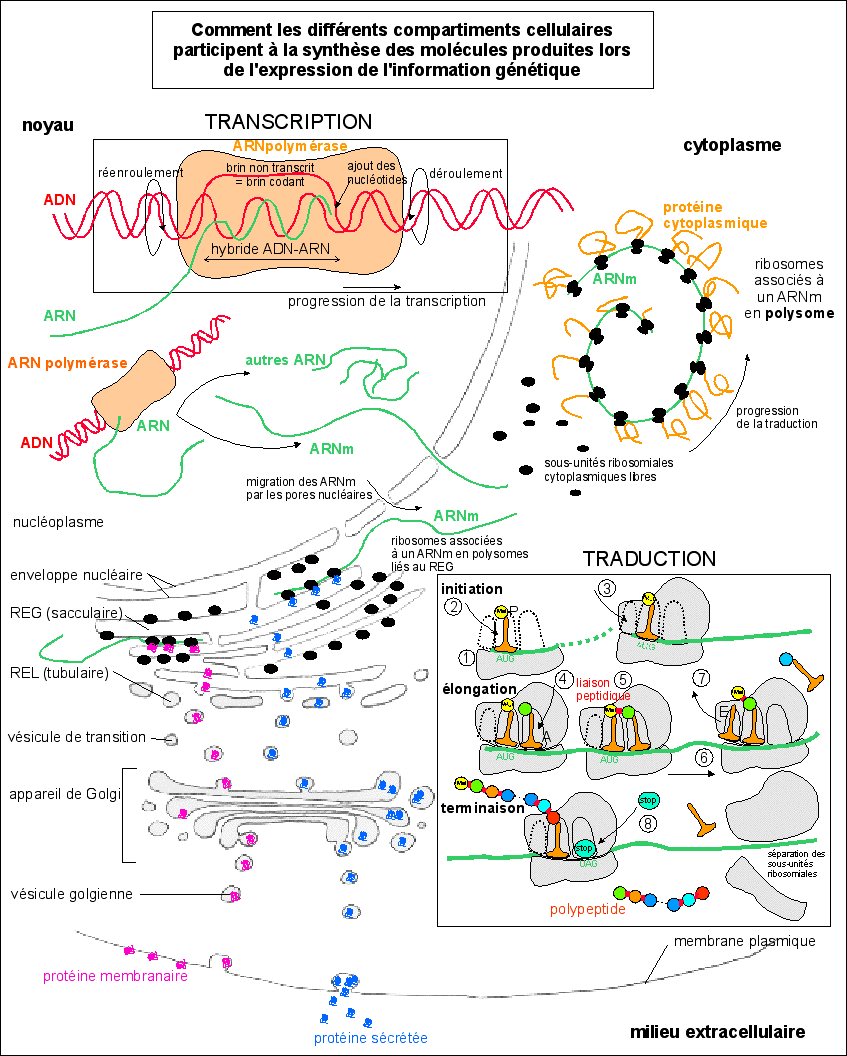
Les étapes de la transcription sont légèrement
plus détaillées que l'exige le strict respect du
programme: cependant seuls les ARNt formant des complexes avec les aa
transportés sont représentés, ce qui me
paraît indispensable à la compréhension de la
traduction; pour éviter toute polémique stérile
vous pouvez les appeler "ARN adaptateur-transporteur d'aa". Les
étapes de la traduction sont les suivantes: 1 - assemblage des
2 sous-unités ribosomiales autour de l'ARNm; 2 -
arrivée et mise en place d'un aa (transporté par son
ARN adaptateur-transporteur) qui correspond selon le code
génétique au codon de l'ARNm placé à la
base du premier site de fixation des ARN transporteurs-adaptateurs
dans le ribosome; on notera que le premier aa est toujours une
méthionine (Met) car le premier codon (initiateur) est
toujours AUG; 3 - arrivée d'un autre aa transporté par
un ARN adaptateur-transporteur dans le second site ribosomial; 4 -
établissement d'une liaison peptidique entre les deux aa
accolés; 5 - déplacement du ribosome d'un codon le long
de l'ARNm; 6 - ce qui implique le départ de l'ARN transporteur
adaptateur qui se détache du peptide en cours de formation et
un changement de site ribosomial pour l'autre ARN
transporteur-adaptateur qui porte la chaîne peptidique en
formation; 7 - après de nombreux cycles d'élongation,
lorsque un codon STOP (UAG, UGA, UAA) devient accessible dans le
second site ribosomial, un complexe vient se fixer sur ce codon et
provoque la terminaison de la traduction, c'est-à-dire la
libération de la chaîne peptidique et de l'ARN
transporteur-adaptateur et la séparation des
sous-unités ribosomiales.
c. Les peptides exportés subissent une maturation dans le
REL puis dans l'appareil de Golgi avant d'atteindre la membrane ou
d'être sécrétés par exocytose
Les peptides synthétisés dans le cytoplasme sont
directement utilisés par la cellule pour le métabolisme
(dans des enzymes...) ou pour des structures
(histones...) (voir tableau
ci-dessous).
Les peptides synthétisés à la surface du REG
migrent dans le REL (reticulum endoplasmique lisse, qui est
tubulaire) puis dans l'appareil de Golgi et sont exportés dans
des vésicules soit vers le membrane (protéines
membranaires) soit vers l'extérieur de la cellule
(protéines sécrétées par exocytose).
C'est par exemple dans l'appareil de Golgi que sont
accrochés les sucres (résidus glucidiques) des
glycoprotéines.
d. L'information génétique est une information
linéaire pour une molécule
La génétique est la science des gènes
(moléculaires et
héréditaires).
La génomique est la science des gènes
moléculaires (dont l'ensemble constitue le
génome).
La protéomique est la science des gènes
moléculaires associés à la synthèse d'une
protéine (dont l'ensemble constitue le
protéome).
Un gène moléculaire est défini par sa
nature chimique (ADN), sa séquence (suite de
monomères) et sa fonction (servir de copie lors de la
transcription). Si l'on considère que les gènes
moléculaires contiennent une information, cette information
est linéaire. De plus, comme la seule fonction de l'ADN connue
précisément est une fonction passive: servir de
modèle lors de la transcription, l'information
génétique est donc une information pour une
molécule (d'ARN).
L'expression de l'information génétique stable de
l'ADN passe d'abord par l'ARN puis peut passer par un
polypeptide.
Par extension on peut aussi dire que l'information
génétique comprend la séquence des
protéines traduites à partir des ARN.
Comme on sait que l'ARN peut être rétrotranscrit en ADN
l'information instable de l'ARN peut être stabilisée
dans l'ADN.
Les gènes
moléculaires sont donc des
segments d'ADN
contenant une information
linéaire exprimée dans une
molécule de type ARN lors de la
transcription
puis, le plus souvent, dans une molécule peptidique
lors de la
traduction.
Ce sont des unités fonctionnelles de l'information
génétique.
L'information
génétique est la séquence de
l'ADN ou de l'ARN ou encore des protéines.
Le contrôle de l'expression de l'information
génétique peut se faire au niveau de la
transcription ou de la traduction.
|
Le nombre de gènes moléculaires des
différentes cellules vivantes n'est pas
précisément connu. Je vous rappelle que
séquencer la totalité de l'ADN d'une cellule ne veut
par dire séquencer le génome (ensemble des
gènes) car on n'est pas du tout sûr que tout l'ADN (des
eucaryotes surtout) est organisé en gènes
moléculaires. On estime à 2000 ou 4000 le nombre de
gènes moléculaires d'Escherichia coli, 6340
celui de Saccharomyces cerevisiae, 10.000 celui des cellules
de Drosophila melanogaster et peut-être 100.000 ou
50.000 pour l'homme. Un gène moléculaire peut
correspondre à plusieurs produits et inversement un produit
peut nécessiter plusieurs gènes moléculaires,
les gènes moléculaires peuvent se chevaucher... bref,
ceci est une autre histoire: celle de la génomique
(science des gènomes) ou biologie moléculaire du
gène moléculaire (avec sa nouvelle branche: la
protéomique qui s'intéresse aux seuls
gènes moléculaires contenant une information pour des
protéines) et elle ne fait que commencer (voir
histoire de la
génétique).
Malgré le séquençage complet du génome
humain on n'espère au mieux que 25.000 gènes
moléculaires associés à des protéines. Si
on ajoute les quelques 2.000 gènes moléculaires
associés à des ARN non traduits cela fait un peu court
pour que l'imaginaire continue de voir dans les protéines
autre chose qu'un support matériel de la vie. Quand au
"pouvoir magique" du gène moléculaire il s'estompe
petit à petit.
Remarque:
Certains bioinformaticiens, cherchant à chiffrer le niveau de
complexité du vivant, considèrent qu'il y a encore 7
ordres de grandeur à atteindre avant de pouvoir rendre compte
du niveau cellulaire (génomique
comparative, interactions rigides, génome, métabolisme
et signalisation unicellulaire, interactions flexibles, complexes
protéiques, métabolisme et signalisation
pluricellulaires, interactions des complexes
protéiques). À mon avis le
problème est pris à "rebrousse poil". La vie, dans sa
complexité, peut être supportée par des formes
simples dans des espaces de contrôle multidimensionnels. Il
semble nécessaire de cesser de raisonner dans un espace
à deux dimensions avec une infinité de formes. La
théorie des modèles (géométrique) de
René Thom permet de complexifier l'espace pour simplifier les
formes. Ce qui est moins intuitif mais très
compréhensible au moins jusqu'à la dimension
4.
|
|
ADN dont la fonction semble
connue
|
ADN dont la fonction
est inconnue
|
|
protéome
|
ADN transcrit (en ARNm) et traduit
en protéines
|
1,2%
|
|
|
régions de contrôle
|
qq %
|
|
|
introns* des gènes protéiques
|
|
31%
|
|
régions non traduites des ARNm (UTR)
|
|
0,7%
|
|
ADN transcrit non traduit (ARN non
ARNm; ARN ribosomiaux, de transfert, petits
ARN et microARN...)
|
0,05%
|
|
|
ADN satellite**
|
|
6,5%
|
|
ADN intergénique
|
|
60%
|
|
TOTAUX estimés
|
~ 10%
|
~ 90%
|
Le génome humain (au sens large
de la totalité de l'ADN nucléaire d'une cellule
humaine) de 3,1 milliards de paires de bases contiendrait
20.000 à 25.000 gènes
protéiques....(Pour la Science, dossier
n°46, 2005, p 18)
* les introns sont des
séquences d'ADN transcrites des gènes protéiques
mais qui sont coupées lors de la maturation des ARNm et ne
participent pas à la formation de l'ARNm qui est
traduit.
|
ADN non
répété
49,5%
|
|
ADN
répété
50,5 %
|
ADN satellite** 6,5
%
|
|
éléments
transposables
44 %
|
information
génétique sous forme d'ADN
|
transposons***
|
3 %
|
|
information
génétique sous forme d'ARN
gène d'une
transcriptase inverse
intégré à
l'élément
|
rétrovirus
endogènes****
|
8 %
|
|
autres
|
33 %
|
** l'ADN satellite sont des
régions répétées des millions de fois de
quelques centaines de paires de nucléotides; elles sont
souvent localisées près du centromère du
chromosome mais peuvent occuper un bras entier. Ces séquences
sont variables entre individus, même d'espèces voisines.
Si l'ADN satellite représente 6,5% de l'ADN humain, 50% de
l'ADN humain est répété. Les
éléments transposables représentent ainsi
44 % de l'ADN ; on y trouve pour 3% des transposons***
(fragments d'ADN qui se déplacent directement dans l'ADN en
s'intégrant ou en se coupant), pour 8 % des
éléments possédant des LTR (longues
répétitions terminales) aux deux
extrémités et qui se comportent comme des
rétrovirus (car ce sont des séquences d'ADN qui
contiennent le gène d'une transcriptase inverse et qui se
déplacent dans l'ADN par l'intermédiaire d'une forme
ARN retrotranscrite) - on les qualifie de rétrovirus
endogènes**** - et pour 33% d'autres éléments
qui contiennent aussi une transcriptase inverse. La plupart des
élément transposables sont non fonctionnels. On
pense que ce sont les vestiges de gènes ou de séquences
qui se sont déplacés chez des ancêtres plus ou
moins lointains.
% des
différentes fonctions
propres* des protéines trouvées
à ce jour - 2001
(in Analyse
génétique moderne, De Boeck,
2001)
|
|
fonctions
|
chez
l'homme
|
chez une plante
Arabidopsis thaliana
|
fonctions
|
|
réplication, transcription,
traduction
|
22
|
15
|
transcription
|
|
régulation des gènes
à partir de signaux extra et
intracellulaires
|
12
|
3
|
traduction
|
|
5
|
tri et stockage des
protéines
|
|
8
|
transduction des signaux
extra-cellulaires
|
|
division cellulaire
|
12
|
6
|
croissance et division
cellulaire
|
|
métabolisme
|
17
|
22
|
métabolisme
|
|
2
|
transport
|
|
2
|
trafic intracellulaire
|
|
10
|
métabolisme secondaire
|
|
6
|
production d'énergie et de
matière organique (dans les mitochondries et les
chloroplastes)
|
|
défense
|
12
|
13
|
maladies et défense
|
|
structure
|
17
|
8
|
structure cellulaire
|
|
inconnue
|
8
|
|
|
|
* les fonctions propres ou locales
désignent des fonctions qui peuvent être
explorées expérimentalement et qui reposent
sur interactions moléculaires (qui en sont la
cause matérielle au sens d'Aristote - voir
annexe);
elles s'opposent aux fonctions participatives ou
globales (non locales) rendant compte de la participation
des protéines au travail cellulaire
(désigné par les trois fonctions globales du
vivant: nutrition, relation et reproduction - ce qui se
rapporte à une cause efficiente au sens d'Aristote)
(voir
cours de seconde).
|
Si les gènes moléculaires n'ont qu'une seule fonction
connue, les protéines en ont beaucoup.. un aperçu.
Remarque: Un article à lire: Les
protéines, rouages des cellules, H. Berman, D. Goodsell et
P. Bourne, Pour la Science, Dossier n°46, janvier mars
2005, p 28-33).
Il y a bien plus dans le matériel génétique
que la seule information génétique au sens
d'information pour une molécule. Nous verrons cela dans le
chapitre 3 après avoir traité des enzymes.
2.2 Un exemple de molécules
fonctionnelles: les protéines enzymatiques
Si l'on trouve encore parfois enzyme au masculin l'usage en fait
un nom féminin: une enzyme.
Les hypothèses concernant le fonctionnement enzymatique seront
présentées ici comme des données puisque le
temps et le programme ne nous permet pas d'en faire l'étude
expérimentale. Ce qui importe ici c'est d'étudier des
produits de l'expression de l'information génétique. Il
ne faut pas considérer ce chapitre comme un cours de biologie
sur les enzymes, ce qu'il n'est
pas.
2.2.1 Les enzymes sont des
biocatalyseurs: elles catalysent des
réactions biochimiques (du vivant) qui appartiennent au
métabolisme
Les enzymes sont des biocatalyseurs (catalyseurs de
réactions biochimiques c'est-à-dire appartenant au
métabolisme) . Ce sont soit quelques rares acides
nucléiques (RNA ou ribozymes) soit, pour la quasi
totalité des enzymes, des protéines.
Elles ont une haute spécificité de substrat:
elles agissent sur un substrat souvent spécifique mais
rarement unique: deux substrats (spécifiques) sont souvent
nécessaires car les réactions chimiques du vivant sont
généralement couplées.
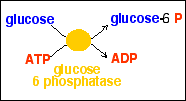
L'enzyme glucose 6 phosphatase
catalyse deux réactions couplées:
- une phosphorylation du glucose
(ajout d'un groupement phosphate sur le carbone n°6 du
glucose)
- et une déphosphorylation de
l'ATP (rupture de la très énergétique
dernière liaison phosphate de l'ATP).
L'énergie fournie par la
déphosphorylation (réaction exothermique) est
supérieure à celle nécessaire pour la
phosphorylation (réaction endothermique). Cette
réaction est essentielle dans toutes les cellules qui
consomment du glucose pour leur fermentation ou leur respiration car
c'est par elle que commence toute utilisation
énergétique de cette molécule (voir la glycolyse
dans le cours de spécialité de Terminale
S).
Elles sont une haute spécificité d'action:
elles accélèrent des réactions chimiques
spécifiques.
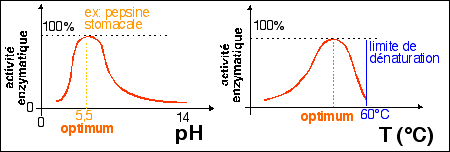
Elles agissent dans les conditions du vivant: en
présence d'eau - et à fortiori in vitro en solution,
même si ce n'est pas leur mode habituel d'action
(voir page
sur la cellule en travaux) - et
dans des conditions de température et de pH très douces
(optima situés bien sûr dans les conditions du
vivant).
Les enzymes sont souvent regroupées structurellement en
complexes enzymatiques, liés au membranes biologiques
ou libres dans le cytoplasme, ce qui accélère
grandement le passage des métabolites d'une enzyme à
une autre (le produit de l'une est le substrat de l'autre et ainsi de
suite, le long de chaînes enzymatiques).
Les enzymes protéiques nécessitent souvent un
cofacteur (un ion comme Fe2+ ou une
métalloprotéine appelée coenzyme).
Pour comprendre la complexité et l'importance des enzymes
ou plutôt de ce que l'on peut appeler les systèmes
enzymatiques, il vous suffit de penser que le chapitre
précédent nous donnait deux magnifiques exemples de
complexes enzymatiques: les ADN polymérases, et les ARN
polymérases, complexes et spécifiques,
contrôlées et extrêmement actives dans la
cellule.
2.2.2 La réaction enzymatique se réalise à
l'intérieur d'une poche appelée
site actif.
L'enzyme est intégralement restituée en fin de
réaction: elle n'est jamais consommée ni
"usée" même si elle peut être
inactivée.
Le site actif peut être décomposé en
site de reconnaissance du substrat (qui peuvent être
multiple) et site catalytique, lieu où est
réalisée la réaction chimique catalysée
(qui est très généralement unique).
L'enzyme se fixe au substrat, catalyse la réaction, puis se
libère des produits.
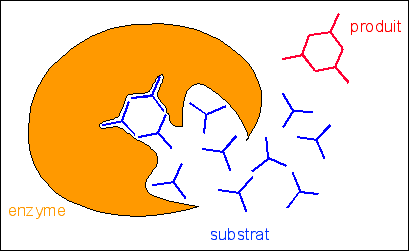
Un schéma de pure
illustration mais faux pour ce qui est de la compréhension
fonctionnelle
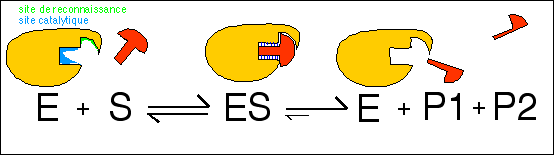
Le site actif peut être
séparé en site de
reconnaissance, qui contrôle la
spécificité de substrat et
site catalytique, qui contrôle la
spécificité d'action.
Ce sont les liaisons faibles
(petits traits bleus) qui fournissent
l'énergie nécessaire à la catalyse.
E = enzyme (restituée intégralement en fin de
réaction); S = substrat; ES = complexe enzyme-substrat; P1 et
P2 = produits de la réaction.
Du point de vue énergétique, l'énergie
fournie par l'enzyme lors de la catalyse est due à
l'établissement de très nombreuses liaisons
faibles au niveau du site actif entre l'enzyme et son substrat
(énergie de liaison).
La cinétique enzymatique étudie IN VITRO
l'évolution de la vitesse de la catalyse enzymatique en
fonction des conditions expérimentales.
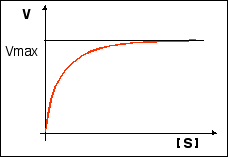
La vitesse V de la réaction enzymatique IN VITRO est
proportionnelle à la concentration en substrat [S]. La
vitesse maximale Vmax est atteinte lorsque l'enzyme est
saturée (tous les sites actifs de toutes les enzymes
présentes en solution sont susceptibles d'être
occupés à une vitesse maximale).
|
Complément
Si le
site
actif à une
forme
complémentaire
du
substrat
(ou d'une partie de celui-ci) - comme une
clé dans une serrure, comment l'enzyme
peut-elle agir - ou la clé
tourner , autrement que par une force
extérieure - par une "main"
?
|
|
Le fait d'imaginer qu'il y a déformation
simultanée de l'enzyme et de son substrat ne change
pas le problème de l'affinité entre l'enzyme
et le substrat...
Il est évident que pour pouvoir fonctionner le
site actif ne doit pas être complémentaire du
substrat mais d'un état de transition entre
substrat(s) et produits. L'image la plus classique
clé-serrure (qui nécessiterait une
main pour tourner la clé) peut être
avantageusement remplacée par celle de la
pièce aimantée-barre
métallique (d'après
Principes de Biochimie, Lehninger, Nelson et Cox,
1994).
Cette analogie reste statique alors que l'enzyme se
déforme habituellement, comme le substrat... la
similitude de conformation entre l'enzyme et l'état
de transition est liée à une forme
dynamique, stable non pas dans R4 (espace
euclidien) mais dans un espace de dimension
inférieure.
|
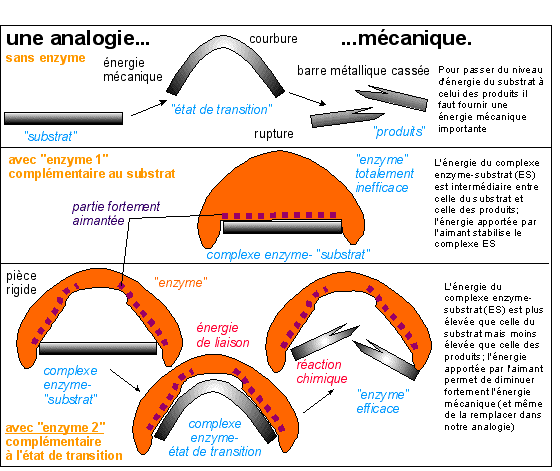
|
|
Une réaction chimique catalysée par une
enzyme permet de "sauter" plus facilement la barrière
d'activation en abaissant son niveau par la formation d'un
complexe enzyme-substrat. En dernier ressort
l'énergie fournie par l'enzyme vient des liaisons
faibles qu'elle établit avec le substrat.
|
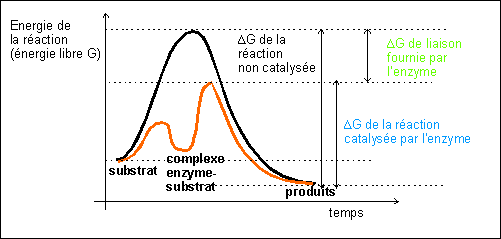
|
|
On est donc amené à proposer une autre
présentation de l'équation de la
cinétique enzymatique qui tienne compte de
l'état de transition (S' - non observable) et non
plus du seul substrat S. ES' étant le
complexe «enzyme -état de
transition».
Pour une réaction chimique présentant un
état de transition S' (très instable) on peut
écrire les deux équilibres:
S
< = >
(S')
< = > P1 +
P2
Ce qui, en présence d'enzyme devient:
E +
S < =
>
ES'
< = > E + P1 +
P2
|
E
+
3S
< = >
E3S'
< = >
E
+
P
Une illustration du rôle de
"moule" du site catalytique (fonction
topologique) : formation d'un complexe "enzyme
-état de transition"
(d'après Encyclopedia Universalis,
catalyse enzymatique - illustration de l'article
"liaisons biochimiques faibles").
|
Remarques:
* Les enzymes sont classées en fonction des réactions
qu'elles catalysent.
Leur nom courant est souvent formé du nom du substrat auquel
on ajoute le suffixe -ase (exemple l'uréase catalyse
l'hydrolyse de l'urée). Mais un système international
de dénomination est de classification a été
adopté répartissant les enzymes protéiques en 6
classes: 1- les oxydoréductases, 2 - les transférases,
3 - les hydrolases, 4 - les lyases, 5 - les isomérases, 6 -
les ligases.
* La réaction enzymatique peut être
inhibée par la présence de molécules qui
l'inactivent ou diminuent sont activité.
Un inhibiteur compétitif ne modifie pas la vitesse de
la réaction enzymatique en présence de son substrat
mais diminue l'affinité apparente de l'enzyme avec son
substrat lorsqu'il est présent en solution. On
interprète son action en proposant que l'inhibiteur
compétitif entre en compétition avec l'enzyme en se
fixant sur la partie de reconnaissance du site actif sans pouvoir
être transformé. Selon son affinité il peut
diminuer plus ou moins l'activité enzymatique. Une comparaison
amusante que je tiens de mes années de classes
préparatoires: l'enzyme est comparée à un
mangeur de lentilles. Chaque cuillerée portée à
la bouche puis avalée est une réaction enzymatique
réalisée. Si l'on ajoute quelques PETITS cailloux dans
le plat de lentilles, le mangeur porte à la bouche des
cuillerées de lentilles avec une belle
régularité mais doit recracher certaines
cuillerées, comportant un petit caillou, ce qui diminue
grandement la vitesse de réaction, mais pas la vitesse avec
laquelle il porte les cuillerées à la bouche... Le
petit caillou joue le rôle d'inhibiteur compétitif.
Un inhibiteur non compétitif ne modifie pas
l'affinité apparente de l'enzyme avec son substrat mais
uniquement la vitesse maximale de la réaction. On
interprète cette action comme le résultat d'une
fixation de l'inhibiteur non compétitif sur un autre site que
le site catalytique de l'enzyme. Pour reprendre ma comparaison avec
le mangeur de lentilles, l'inhibiteur compétitif peut
être comparé à un GROS caillou dans les
lentilles. Le mangeur les voit et les évite, ce qui diminue
singulièrement sa vitesse de rotation des
cuillerées.
L'interprétation que l'on fait de l'action des inhibiteurs va
de pair avec celle des analogues de structure qui sont des
substances voisines (au sens topologique) de molécules
métaboliques ou informationnelles habituelles mais qui
bloquent ou diminuent la fonction des molécules fonctionnelles
ou informatives auxquelles elles se lient. La mise au point
d'analogues de substrat est une des étapes essentielles de la
recherche pharmaceutique.
2.2.3 La fonction enzymatique est
une "fonction topologique"
Je rappelle que le mot fonction a tout
intérêt à être défini au sens
mathématique de la façon la plus rigoureuse qui soit.
Une fonction biologique c'est un phénomène qui peut
être représenté par une fonction
mathématique (voir cours
de seconde et
une petite page
sur les mathématiques en SVT en
seconde). Ici nous nous
intéressons à des fonction locales et donc que
l'on peut explorer expérimentalement.
La topologie est une branche des
mathématiques qui s'intéresse aux
propriétés de l'espace et des ensembles de
définition des fonctions du seul point de vue qualitatif; elle
précise les notions de
continuité-discontinuité, bords,
limites...
Être une enzyme signifie avoir une fonction
enzymatique. C'est-à-dire une fonction catalytique
(accélération d'une réaction
chimique sans altération du catalyseur). On pourrait
dire que cette fonction est topologique dans le sens
où elle repose sur une similitude de forme entre l'enzyme et
un état transitionnel du(des) substrat(s).
Mais comment comprendre
l'activité enzymatique ? Une fois encore on a (au moins) deux
attitudes (voir aussi la page de synthèse sur
les niveaux
d'organisation du vivant) :
- chercher à comprendre comment est
"contrôlée" la forme de l'enzyme (et plus
particulièrement du site actif) et en quoi cette forme permet
l'activité catalytique au niveau chimique (liaisons faibles,
transfert de charges...). C'est la voie du
réductionnisme. Je vous propose une autre voie.
- accepter comme un fait expérimental (empirique) ce
contrôle de la forme et chercher à en formaliser les
limites. C'est la voie du structuralisme. Toute fonction
topologique - qui repose sur une correspondance des formes - pourrait
être appelée enzymatique. René Thom en a
dessiné les contours (dans Modèles mathématiques
de la morphogénèse (1971, 1974, 1980) notamment).
Voici quelques exemples de ces deux attitudes:
2.3 Du phénotype au génotype
et retour
Les éléments nécessaires
à la compréhension de cette partie sont un
complément du cours
de seconde et sont
détaillés dans une page
annexe sur les phénotypes et
génotypes.
2.3.1 Un peu d'histoire: : l'embrouille
héréditaro-moléculaire
L'histoire commence en 1902
avec la définition des gènes
(héréditaires), des
allèles, du
génotype
(héréditaire) et du
phénotype
(héréditaire) chez les EUCARYOTES.
L'histoire semble se simplifier avec
l'avènement d'une biologie moléculaire des
années 1960 fondée sur le rôle de
l'ADN dans la synthèse des protéines principalement
à partir de travaux sur les PROCARYOTES - au début du
moins. Les termes de gène
moléculaire,
"allèle",
"génotype" et
"phénotype" sont
redéfinis dans une vision globale du vivant.
Mais près de un demi-siècle plus tard ces deux visions
ne spont toujours pas unifiées. Il y donc vraiment
un fossé entre ces deux
visions.
Le problème peut être résumé en une
remarque: il n'y a pas de prédiction du phénotype
par le génotype (en utilisant bien sûr
l'analogie moléculaire et donc les sens de phénotype
moléculaire et de génotype moléculaire). On peut
aussi le formuler en affirmant que ce qui s'applique aux Procaryotes
ou aux eucaryotes unicellulaires ne s'applique pas simplement aux
pluricellulaires.
On peut donc supposer une erreur de méthode (du discontinu au
continu). C'est ce qui a fait redémarrer les études
biologiques intégratives et la physiologie quelque peu
abandonnées au profit de la biologie moléculaire.
Voici un tableau qui résume ces deux
visions (voir aussi cours
de seconde):
Le but n'est pas
d'embrouiller un élève mais l'aider à comprendre
ce qui n'est pas dit par l'auteur d'un texte qui emploie des mots
avec un sens caché...
|
gène
|
Un gène
héréditaire est une portion de
chromosome associée à la transmission d'un
caractère héréditaire (uniquement chez
les eucaryotes)
|
Un gène
moléculaire est un segment d'ADN transcrit en
ARN et éventuellement en protéine (aussi bien
chez les procaryotes que chez les eucaryotes)
|
|
allèle
|
Un allèle est la
forme d'un gène
héréditaire.
|
 AUCUN sens analogique légitime
AUCUN sens analogique légitime
(ne pas employer
"allèle" pour désigner une séquence
d'un gène moléculaire)
|
|
|
définition HISTORIQUE claire
|
définition analogique et étendue
souvent abusive
|
|
génotype
|
Le génotype d'un
gène héréditaire est l'ensemble des
allèles de ce gène.
|
Le génotype
moléculaire d'un gène moléculaire
est constitué analogiquement par les
séquences de ce gène
moléculaire.
|
|
Le génotype
héréditaire d'un organisme est l'ensemble
des allèles de tous les gènes
héréditaires de cet organisme*.
|
Le génotype
moléculaire d'une cellule (ou d'un organisme) est
constitué par extension par toutes les
séquences de tous leurs gènes
moléculaires**.
|
|
phénotype
|
Le phénotype d'un
gène héréditaire c'est l'ensemble
des caractères visibles associés à la
possession de ce gène.
|
Le phénotype
moléculaire d'un gène moléculaire
est constitué analogiquement par l'ensemble des
produits (ARN et peptides) résultants de sa
transcription et éventuellement de sa
traduction.
|
|
Le phénotype
héréditaire d'un organisme c'est
l'ensemble des caractères visibles
associés à la possession de tous ses
gènes héréditaires*.
|
Le phénotype
moléculaire d'une cellule (ou d'un organisme) est
constitué par extension par l'ensemble tous les
produits (ARN et peptides) résultants de sa
transcription et éventuellement de sa traduction de
TOUS leurs gènes moléculaires.**
|
|
* il est clair que cette vision des
choses n'est pas fermée; toutes les
caractéristiques d'un organisme ne sont pas
explicables par des gènes héréditaires.
Bien au contraire. Seules quelques caractéristiques
(et peut-être quasiment aucune chez l'homme) sont
susceptibles d'être expliquées par une
transmission chromosomique simple telle qu'elle avait
imaginée par Morgan (voir cours
de spécialité de
TS).
|
** Si cette définition est
relativement utilisable pour un procaryote ou pour un
unicellulaire eucaryote, elle n'a pas beaucoup de sens pour
un eucaryote pluricellulaire pour qui le nombre de
gènes moléculaires est loin d'être
connu, pour ne rien dire de l'homme. Des cellules de
même génotype moléculaire peuvent avoir
des phénotypes moléculaires différents.
|
|
Sans précision autre les
termes de génotype et de
phénotype d'un gène ou
d'un organisme devraient être considérés
dans leur acception héréditaire avant de
suspecter une acception moléculaire, qui reste
souvent abusive.
|
Remarques:
* Cependant il ne faut pas "jeter le bébé avec l'eau du
bain". Non pas abandonner l'hérédité
chromosomique mais la redécouvrir avec des théories qui
ne ne se basent plus sur la vision simpliste d'un
molécularisme (qui n'existe plus dans son expression
caricaturale). On peut par exemple lire l' « Avant propos »
de Michel Morange, Pour la Science, Dossier n°46, janvier-mars
2005, p 2-3 pour voir que derrière l'abandon de la vision
simpliste, on rechigne encore à se lancer dans une
véritable biologie théorique "à la René
Thom".
* au sujet de la notion de phénotype alternatif.
Ce terme n'apparaît à ma connaissance que dans les
documents d'accompagnement du programme (tapez-le sous Google...) et
est repris par le Nathan notamment; il est définit
officiellement de la façon suivante : « les
phénotypes alternatifs sont les variations d'un même
caractère présentées par divers individus de la
même espèce ». Si l'on s'entend sur la
notion de caractère héréditaire (sous-entendu
mendélien ou morganien), un phénotype alternatif c'est
un allèleh, forme (ou variation) d'un gène
héréditaire, support d'un caractère
héréditaire. Il est intéressant car, bien
qu'inemployable, il montre que ses concepteurs ont vu qu'il y
avait un problème insoluble si l'on choisissait de nommer
"allèle", la séquence d'un gène
moléculaire. Si toute caractéristique est
phénotypique, tout phénomène (sauf l'ADN) est
phénotype: on revient à la stricte étymologie.
Mais alors le mot ne signifie plus rien car l'ADN est aussi
phénotype. Il me paraît essentiel de dire que le terme
phénotype n'a de sens que dans le cadre d'une théorie
héréditaire.
* l'épigénétique
désigne chez les généticiens "ce qui modifie le
génétique" (au sens de support de l'information
génétique, c'est-à-dire l'ADN ou l'ARN) et vient
de la cellule ou d'une autre cellule: les biologistes
moléculaires désignent ainsi une information
postgénétique, reposant principalement sur des
méthylations de l'ADN et le rôle des histones dans
l'expression de l'ADN. On trouve ainsi récemment dans la
littérature le terme d'épigénome. Mais
pourquoi limiter l'épigénétique au
postgénétique ? Dans le cadre d'une
génétique moléculaire, celle des procaryotes,
TOUT EST GÉNÉTIQUE car le génétique est
assimilé à la synthèse des protéines
(mais peut-être pas de toutes) que l'on trouve partout dans la
matière vivante. Comme les gènes moléculaires
sont eux mêmes dans un cytoplasme cellulaire, lui-même
dans un environnement extracellulaire : le fonctionnement des
gènes est épigénétique et donc TOUT EST
ÉPIGÉNÉTIQUE. Belle avancée
théorique. Historiquement, à ma connaissance, ce terme
renvoie d'une part à celui d'épigénèse
(épigénétique est alors équivalent
d'épigénésique -
l'épigénèse étant un processus de
transformation progressive de l'être vivant au cours du
développement par opposition au préformisme (ou
évolutionnisme, dans le sens ancien, voir deux articles sur
une page de
textes sur l'évolution). D'autre
part, mais toujours dans ce sens de lié au
développement, le terme d'épigénèse se
superpose assez facilement a celui d'acquis par opposition à
l'inné; on le retrouve notamment dans les travaux faisant
référence au développement psychique de l'enfant
(Piaget, Freud) ou la neurologie d'une façon plus
générale. Enfin, et d'une façon encore
différente, on peut citer la notion de "paysage
épigénétique" due a Waddington (voir
page
annexe). Sans prétendre faire
œuvre d'historien je crois que l'on peut dire que les
différents sens récents
d'épigénétique se recoupent comme étant
une référence au modèle
épigénétique du développement,
actuellement dominant. (retour
tableau - info
génétique)
2.3.2 Des espoirs déçus aux espoirs naissants
2.3.2.1 - Sémiophysique de
l'ADN (partie culturelle en
vert)
La sémiologie est
l'étude du sens (étymologiquement l'étude des
signes: du grec "séméion" = "signe"). L'ADN a
souvent été considéré comme un texte dont
le sens est à déchiffrer. Or, la sémiologie a
pris deux directions qui peuvent s'appliquer à l'étude
du sens de l'ADN (voir article
"signe et sens" de Paul Ricœur dans
l'EU):
- Le sens le plus courant est ce que l'on appelle le sens
explicatif d'un texte. Dans cette vision, la signification de
l'ADN est rapportée à sa structure (linéaire !);
le sens de l'ADN, c'est son organisation en unités
fonctionnelles: les gènes, les unités
régulatrices, les éléments transposables... On
peut dire que cette explication marque actuellement le pas. Chez
l'homme il n'y a que 25.000 gènes moléculaires
(associés à des protéines) connus en ce
début d'année 2005. Les séquences
régulatrices sont de moins en moins nombreuses à
être mises en évidence. Même si des ARN
régulateurs semblent prendre le relais
(en oubliant le travail de
précurseur de Beljanski),
une part croissante d'ADN semble dénué de tout sens. Le
terme d'ADN poubelle fait recette pour désigner
des séquences non fonctionnelles mais très voisines de
séquences fonctionnelles connues.
- L'analyse sémiologique a débouché sur une
autre approche du sens d'un texte qui est celle connue sous le nom
d'interprétation (du "mouvement du texte ... vers sa
référence ... située au-dehors"; on
recherche par exemple le contexte historique, les destinataires, les
résonances avec d'autres textes...). Le sens
"interprétatif" de l'ADN n'en est qu'à ses
balbutiements. La première approche, que certains qualifient
de déterministe (ou modèle instructif)
est en cours d'abandon. On ne croît plus qu'un gène
moléculaire a un niveau d'expression contrôlé par
des séquences précises situées en amont ou en
aval, qui agissent en fonction des conditions métaboliques de
la cellule en réglant ainsi la concentration cellulaire en une
protéine. Certains
(voir par exemple:
L'expression des gènes : la révolution
probabiliste, Jean-Jacques Kupiec, Pour la Science, dossier
n°46, janvier-mars 2005, p 34-38)
veulent y substituer ce qu'ils qualifient de modèle
darwinien ou sélectif: chaque gène
moléculaire est exprimé de façon probabiliste
dans la cellule en fonction des conditions internes,
elles-mêmes sous la dépendance des informations
extracellulaires qui stabilisent les types cellulaires acquis de
façon aléatoire par les cellules. Cette vision est
intéressante car elle pose le problème de la
causalité (voir les niveaux
d'organisation du vivant) mais elle
reste dans une vision réductionniste
(voir ci-dessus)
qui me paraît avoir beaucoup de peine à passer du niveau
moléculaire au niveau cellulaire. Si l'on veut entrer dans une
vision empiriste-structurale au niveau cytologique on peut par
exemple s'intéresser à la piste donnée par
René Thom: le rôle de l'ADN est à rapporter
à celui des chromosomes, dans une fonction globale
reproductive.
|
René Thom in
stabilite.pdf
p 215 « J'aimerais, quant à moi,
considérer les chromosomes comme des organites en
tout point semblables aux autres et dont le
métabolisme ambiant assure à la fois la
stabilité, la duplication et les éventuelles
variations ; dans le programme général ici
proposé qui consiste à interpréter
dynamiquement les formes de l'ultrastructure cellulaire, on
se heurte à une difficulté bien connue ; nous
ignorons en effet totalement la nature des forces qui
assurent le fonctionnement des organites cellulaires, qui
causent la mitose, la méiose, le crossing-over etc.
Il ne faut pas s'étonner de notre ignorance ; si l'on
songe aux difficultés que rencontre l'étude
mathématique d'une structure condensée aussi
simple que celle décrite par le modèle
d'Ising, (en théorie de la solidification), il n'est
pas étrange que nous ne sachions pas évaluer
l'effet de la présence d'une structure
macromoléculaire sur le milieu ambiant du cytoplasme
vivant et, par réaction, l'effet de ce milieu sur la
structure. C'est pourquoi, on peut tenter une
démarche inverse de celle préconisée
d'ordinaire ; au lieu d'expliquer la morphogenèse
im Grossen par des modifications de l'ultrastructure
cellulaire, expliquer l'ultrastructure cellulaire par des
schémas dynamiques semblables à ceux de la
morphogenèse globale, mais à l'échelle
de la cellule. Certes, ce programme ne peut guère
être abordé sans une part considérable
d'arbitraire dans l'interprétation dynamique des
organites cellulaires ; c'est un danger inévitable
qu'il faut courir dans une tentative aussi neuve.»
p 218 « Quel sens donner alors au message
porté par l'ADN des chromosomes ? Il me faut
répondre ici par une analogie ; on sait, en
géométrie des systèmes
différentiels, qu'en sa presque totalité, la
forme d'un système dynamique structurellement stable,
comme un champ de gradient, est déterminée par
les points singuliers du champ (positions d'équilibre
du système où le champ est nul). Or,
considérons une cellule en voie de division comme un
système dynamique dont la duplication est
structurellement stable ; il existera dans cette duplication
des éléments spectraux, formant l'ensemble des
points où s'initie la duplication spatiale. Or, ce
sont les chromosomes et les molécules d'acide
nucléique qui jouent ce rôle. Toute
modification assez forte de la dynamique d'auto-reproduction
se traduit par un changement de structure
(géométrique ou chimique) de ses
éléments spectraux, de ses
singularités. C'est, je crois, en ce sens et en ce
sens seulement, qu'on peut voir dans l'ADN le support de
l'information génétique.»
|
Le terme de sémiophysique est un néologisme
inventé par Jean Petitot pour désigner une "physique du
sens" et repris par René Thom (je ne cesse de conseiller de se
rapporter à l'article "forme" de Jean Petitot dans l'EU).
|
René Thom,
1968f4.pdf
« Mais ses propres succès ont placé la
Biologie Moléculaire dans une position
épistémologiquement inconfortable :
fondamentalement matérialiste, elle pense que la
structure des êtres vivants n'est qu'un agencement
moléculaire ; comment dès lors expliquer -
dans les mêmes termes d'interaction moléculaire
- la stabilité d'un organisme déjà
énorme à son échelle, comme un corps
bactérien, a fortiori celle d'un Métazoaire
comme la puce ou l'éléphant ? De ce point de
vue, le contraste est grand entre l'exigence
matérialiste de départ, et le langage
outrageusement anthropomorphe (molécules
messagères, codage et décodage d'une
information, démiurgie enzymatique) que
nécessite la description de la dynamique vitale. De
cette contradiction interne, certains spécialistes, -
encore tout imbus de triomphalisme technique - sont à
peine conscients. D'autres ont cru trouver une solution dans
la théorie de l'information, qui, visiblement,
n'était pas faite pour cela. On ne relira pas sans
rire, dans quelques années, les pages où tel
biologiste s'étonne que le génome humain ne
contienne guère plus que mille fois plus
d'informations que celui de l'humble Colibacille, et qu'il
en contienne beaucoup moins que ceux du Triton ou du
Blé... Comme si la distribution des
nucléotides sur la chaîne d'ADN pouvait
être équiprobable ! Comme si l'entretien et la
duplication du matériel nucléique n'exigeaient
pas la présence d'un milieu cytoplasmique qui lui est
étroitement et spécifiquement adapté !
À cet égard, une seule hypothèse
paraît plausible : en raison des contraintes qu'impose
la viabilité globale du système, la
chaîne d'ADN doit s'organiser en segments relativement
autonomes et stables, les segments « significatifs
», qui peuvent d'ailleurs présenter une
hiérarchie de subordinations fonctionnelles, tout
comme notre langage se décompose, en phrases, enmots
et en lettres. Le « code génétique
»ne correspond guère qu'au niveau le plus
élémentaire, celui des lettres dans le mot, le
« niveau de première articulation » des
linguistes. Le génome s'est constitué au cours
de l'évolution par une combinatoire de segments
significatifs (impliquant des redoublements, des
permutations, des séparations topologiques, etc.) qui
simulait la combinatoire des structures de régulation
globale de l'organisme en voie de complexification
progressive.
Bien entendu, une telle théorie ne peut actuellement
qu'être à peine ébauchée. Mais
ceci montre combien il est nécessaire de disposer
d'une théorie qui rétablisse le lien jusqu'ici
manquant entre dynamique globale et morphologie locale. Or
une discipline qui cherche à préciser le
rapport entre une situation dynamique globale (le «
signifié »), et la morphologie locale en
laquelle elle se manifeste (le « signifiant »),
n'est-elle pas précisément une «
sémiologie » ?»
|
2.3.2.2 Des réussites de la biologie moléculaire du
gène moléculaire qu'il faut bien se garder
d'ériger en modèle
a - Drépanocytose et thalassémies: des
anomalies dans la séquence primaire de l'hémoglobine
(une protéine à fer) à l'étude des
séquences des gènes moléculaires des
différentes chaînes de l'hémoglobine dans des
populations atteintes de différentes maladies ou des
maladies à différents niveaux
phénotypiques
|
Interpré-tation
moléculaire
et biologique non
héréditaire
|
niveau moléculaire
|
niveau cellulaire
|
niveau de l'organisme
|
Interprétation
moléculaire et biologique non
héréditaire
|
|
L'hémoglobine est un
tétramère (formée de 4
sous-unités, identiques 2 à 2). Chaque
chaîne est une hétéroprotéine car
elle est composée d'une chaîne protéique
(la globine) et d'un groupement à Fe
(l'hème).
La chaîne ß de l'hémoglobine contenue
dans les hématies falciformes et qui se
polymérise présente une différence avec
la chaîne ß habituelle. L'aa situé en
position 6 est une valine (Val) ou lieu d'un acide
glutamique (Glu).
Ce changement d'un seul aa suffirait à expliquer la
polymérisation de la molécule et la
différence de migration des solutions d'Hb lors d'une
électrophorèse*.
La valine est un aa beaucoup plus hydrophobe que l'acide
glutamique qui est le plus hydrophile des aa
(voir code
génétique).
Ce qui pourrait expliquer que la valine, ne pouvant entrer
aisément dans la partie globuleuse de la
protéine; se retrouverait à
l'extérieur, ce qui pourrait changer grandement la
forme d'une partie de la molécule et favoriser la
polymérisation.
Ce changement d'hydrophilie, associé à la
polymérisation, pourrait aussi expliquer la
différence de migration
électrophorétique (mais
certains éléments sont peu clairs et je
renonce provisoirement à les
élucider...).
Le gène de la chaîne ß chez l'homme
est porté par le chromosome 11.
On considère que le changement d'aa est du à
une mutation ponctuelle (d'un seul nucléotide
dans un seul codon: GAG changé en GUG dans l'ARNm
soit au niveau de l'ADN, CTC changé en CAC, pour le
brin codant (non transcrit) et donc GAG vers GTG pour le
brin non codant (transcrit).
|
Certaines hématies dites falciformes (en
forme de "faux") ou drépanocytaires (en grec
"drepanos"= faux) sont relativement "rigides" et fortement
déformées; elles se bloquent dans les
capillaires.
L'hémoglobine contenue dans les globules
drépanocytaires est polymérisée sous
une forme fibreuse.
Cette polymérisation déforme
l'hématie (en faucille) mais modifie
surtout ses propriétés
viscoélastiques (les hématies forment
habituellement des rouleaux). Aux écoulements lents,
c'est la thixotropie de l'hématie qui est
diminuée (propriété de changement de
phase solide (gel) - liquide).
Vous pouvez lire à ce sujet l'article
sur l'hémorhéologie dans l'EU et vous reporter
à la page
sur l'eau dans la cellule pour
vous persuader de l'importance de l'eau comme
élément structurant du cytoplasme. Oubliez les
tubes à essais et comprenez bien que le cytoplasme de
l'hématie est un milieu "solide" avec des
molécules (d'Hb) entourées d'une fine
pellicule d'eau. Une hématie contient 70%
d'eau, 25 % d'Hb et le reste d'éléments
organiques et d'ions (notamment du potassium).
Les hématies ne contiennent pas de noyau et ont
une durée de vie courte (120 jours). La
synthèse d'Hb a donc lieu pendant leur période
de maturation (érythroblaste avec
noyau puis réticulocyte sans noyau avec
ribosomes) qui dure environ trois jours après
un stade de prolifération (4j) à partir de
cellules souches de la moelle rouge des os. Le sang humain
contient 4 à 5 millions d'hématies par
mm3 (soit environ 25.000 milliards au total) et
en renouvelle 200 milliards par jour. Elles sont
détruites dans le système
réticulo-histiocytaire (ou
endothélial - voir ancien
TP - au niveau du foie, de la
rate et de la moëlle).
|
la
drépanocytose est
une maladie dont les symptômes
(douleurs, fièvre (due à l'éclatement
de globules rouges), vertiges, maux de tête,
anémie (diminution du nombre de globules rouges qui
sont détruits parcequ'anormaux...), lésions
d'organes...) peuvent tous être rattachés
à des problèmes circulatoires au niveau des
capillaires (certains sont même bouchés ce
qui peut se voir par échographie à effet
Doppler).
|
|
L'ensembles de caractéristiques de cette maladie
constitue un tableau clinique que l'on peut qualifier de
phénotype malade (ou drépanocytaire) et
qui s'oppose au phénotype sain. Ces deux notions ne
sont pas équivalentes.
On considère que le malade est atteint d'une
maladie héréditaire associée à
un gène porté par un chromosome. Ce
gène peut donc être sous l'allèle
malade (drépanocytaire - noté S) ou sous
l'allèle sain (noté A). Comme chaque
individu est porteur de deux gènes (l'un paternel,
l'autre maternel) les génotypes
héréditaires possibles (on porte les
allèles de part et d'autre d'une barre de fraction)
sont A//A pour l'homozygote sain, A//S pour
l'hétérozygote (qui présente des
symptômes plus ou moins légers) et S//S pour
l'homozygote malade (qui présente souvent des
symptômes graves).
|
interprétation
héréditaire
|
|
une maladie de
l'hémoglobine
|
une maladie du comportement
rhéologique des hématies
|
une maladie circulatoire
héréditaire
|
|
phénotype
moléculaire
= ensemble des
produits (ARN et peptides) résultants de la
transcription et éventuellement de la traduction d'un
gène moléculaire
|
phénotype
moléculaire au niveau
cellulaire = ensemble des
phénotypes moléculaires permettant d'expliquer
certaines caractéristiques cellulaires (on se limite
aux explications génétiques
moléculaires)
|
phénotype
héréditaire d'un
allèle
= ensemble des
caractères associés à un
allèle
phénotype
héréditaire de l'organisme
= ensemble des tous ses
caractères héréditaires
|
|
phénotype
héréditaire cellulaire
= ensemble des
caractéristiques cellulaires permettant d'expliquer
un phénotype héréditaire
|
Mais:
les hémoglobinopathies (maladies de l'Hb) sont
complexes du fait des nombreuse chaînes impliquées qui
changent au cours de la vie et pour lesquelles des variations sont
possibles (voir ancien
cours de spécialité).
On connaît actuellement près de 600 hémoglobines
différentes dont 476 formes pour la seule chaîne
ß. De plus les chaînes changent en fonction de
l'âge. Les différentes associations possibles de
chaînes et les différentes séquences de ces
chaînes ont donné lieu à des hémoglobines
que l'on sépare en groupes
éléctrophorétiques. Certaines correspondent
à des maladies circulatoires ou respiratoires que l'on
nomme thalassémies (car elles se trouvent surtout dans
les populations situées autour de la
Méditerranée: LA mer des grecs: "thalassa" = mer
en grec).
Donc, ce qui a fonctionné dans le sens phénotype
(héréditaire) vers niveau moléculaire ne peut
pas être utilisé dans l'autre sens, sans
précaution. Il n'y a pas un déterminisme au sens strict
mais un déterminisme et un indéterminisme
(voir page
sur les niveaux d'organisation du
vivant). Pour une
hémoglobine donnée on n'a pas forcément un
phénotype héréditaire associé (visible au
niveau de l'individu et associé à un allèle,
forme d'un gène héréditaire situé sur un
chromosome). Certes tout gène moléculaire
associé à une chaîne d'Hb correspond bien
à une protéine mais cette protéine n'est
peut-être pas synthétisée partout, tout le temps
et ne donne pas forcément lieu à un caractère
observable. Dans ce cas on ne peut pas dire qu'elle correspond
à un phénotype héréditaire. Les
symptômes de la drépanocytose sont principalement dus
à la modification de la rhéologie du globule rouge (et
donc peut-être pas uniquement de la polymérisation de
l'Hb) et non pas au fonctionnement de la molécule d'Hb
fixatrice de dioxygène et de dioxyde de carbone. De même
si dans le cas présent on pense pouvoir expliquer TOUS les
symptômes de la maladie au niveau de l'organisme par des
dérèglements au niveau cellulaire et
moléculaires, il n'est reste pas moins que la maladie reste un
phénomène dont certains aspects sont
indéterminés, ce que l'on appelle par exemple le
terrain biologique ou ce que l'on attribue aux facteurs
environnementaux. Il me paraît donc souhaitable de garder
chaque niveau d'interprétation comme à la fois
dépendant et indépendant des autres niveaux sans
chercher une compréhension définitive.
|
De la connaissance de
différents niveaux d'organisation du
vivant
(<<<<<<<
sens explicatif réductionniste ----- = sens
explicatif structural---------- >>>>>>>
sens prédictif)
|
|
niveau
moléculaire
ou niveau des associations supramoléculaires
(fonctions locales)
|
<<<<
>>
des lois locales
(déterminisme fort dont l'exploration
expérimentale est actuellement en cours de
façon plus qu'intensive...) et beaucoup
d'indéterminisme (seule une description
statistique est alors possible)
|
niveau
cellulaire, premier
niveau global du vivant, chez les unicellulaires, certaines
cellules peuvent ne pas participer de toutes les fonctions
globales
|
<<<
>
un déterminisme fort
(les associations cellulaires en tissus et organes sont
très stables dans le vivant...) mais dont les lois
semblent difficiles à appréhender par
l'expérience; un renouveau pourrait être
apporté par une modélisation
géométrique
|
niveau de
l'organisme, niveau
global principal, l'organisme doit présenter toutes
les fonctions globales
|
|
=
|
=
|
=
|
|
Une myriade de configurations
moléculaires
|
des types cellulaires
stables en nombre limité - c'est le niveau qui a le
plus besoin de mathématisation
(géométrisation)
|
des individus très
stables et résistants aux perturbations mais
adaptables et évolutifs... un
mystère.
|
|
pour chaque niveau on peut
définir des paramètres (qualitatifs ou
quantitatifs) pour lesquels il est nécessaire de
préciser le domaine de définition: un
paramètre moléculaire peut avoir un domaine de
définition très large (la présence d'un
marqueur membranaire du groupe CMH peut par exemple
concerner la plupart des populations cellulaires) ou plus
restreinte (la molécule d'Hb n'est
synthétisée que par certaines cellules de la
lignée myéloïde). De la même
façon l'hérédité peut être
définie à chaque niveau. Le niveau
moléculaire n'est cependant pas le niveau le plus
facile et ce n'est pas pour rien que la théorie
historique de Mendel, complétée par Morgan, a
d'abord été définie au niveau de
l'organisme. L'extension que l'on essaie parfois de
réaliser au niveau moléculaire ne garde son
pouvoir explicatif que dans de rares cas que l'on peut
désormais considérer comme l'exception. Il est
temps de trouver une autre théorie de
l'hérédité qui se décline aux
différents niveaux d'organisation.
|
*
Pour l'histoire (in EU) on peut encore
signaler que « c'est Linus Pauling (encore lui !!!) en
1949 qui avait mis en évidence une migration
électrophorétique anormale de l'hémoglobine des
personnes drépanocytaires. (...) Vernon Ingram eut
l'idée de fragmenter les hémoglobines de sujets normaux
et de patients atteints de drépanocytose par une enzyme (la
trypsine) puis de comparer le comportement migratoire des peptides
ainsi obtenus. Ni en électrophorèse ni en
chromatographie, on n'observait de différence. Mais, en
combinant les deux approches (voir Nathan), et en effectuant la
chromatographie perpendiculaire à
l'électrophorèse, Ingram mit en évidence le
comportement identique de tous les peptides sauf un :
hémoglobine A (normale) et hémoglobine S
(drépanocytaire) ne différaient que par un acide
aminé (acide glutamique substitué par une valine en
position 6 de la chaîne b-globine). Cette découverte fut
le point de départ de recherches menées par de
nombreuses équipes et étalées sur des
années. Un peu plus tard, avec un étudiant, John Hunt,
Ingram détermina aussi la substitution responsable d'une autre
hémoglobine anormale (hémoglobine C). (...) On lui doit
aussi le schéma évolutif phylogénique des
gènes de globine.» retour
b - Les groupes sanguins de Landsteiner (ABO) : un autre
exemple de liaison entre le phénotype (de 3 allèles, 2
allèles codominants A et B et un allèle récessif
o) et les gènes moléculaires associés à
des enzymes de type glycosytransférase (agissant au niveau de
l'appareil de Golgi) intervenant dans le synthèse de
chaînes glucidiques associées à des
protéines ou à des lipides membranaires. Les enzymes
accrochent un résidu glucidique à une substance H,
elle-même glucidique . L'enzyme A ajoute le
N-acétylgalactosamine , et l'enzyme B le D-galactose; o
correspondant à une enzyme non fonctionnelle.
La présentation simpliste de
cet exemple commence à dater fortement. D'abord parce que
l'hémotypologie a fait des progrès étonnants
notamment avec la découverte de très nombreux variants
(A1, A2, A3, Ax,
Am, B, H, Le, Se pour le seul groupe ABO), sachant que le
nombre de groupes est maintenant supérieur à la
dizaine. Ensuite du fait de la présence de ces mêmes
marqueurs sur d'autres types cellulaires (leucocytes (globules
blancs) et plaquettes; cellules conjonctives et nerveuses....) ou
même dans certaines sécrétions (sueur, sperme,
salive...), ce qui permet des recherches de groupe sanguins... sans
trace de sang (en criminologie par exemple). Enfin parce que la
génétique de ces marqueurs cellulaires est
extrêmement complexe: remaniements de gènes
moléculaires incessants, épissage alternatif et autres
mécanismes qui empêchent tout pouvoir prédictif
simple de la connaissance des "génotypes" au sens de la
biologie moléculaire.
L'idée marquante est encore une fois que si la
génétique mendélienne peut rendre compte de la
transmission héréditaire simple de ces
caractères sanguins, c'est qu'il existe probablement un
mécanisme stabilisateur qui se situe à un autre niveau
que le niveau génomique. L'extrême variabilité
individuelle moléculaire convergeant vers une
stabilité de la fonction qui est justement ce que cherche
à exprimer de façon mathématique une
théorie biologique.
c - L'alcaptonurie: un exemple de passage réussi
entre les symptômes liés à une maladie et une
cause enzymatique reliée à des anomalies dans des
gènes moléculaires associés à des
enzymes. (en travaux)
d - La phénylcétonurie: (en travaux)
e - L'enzyme glucose-6-phosphate déshydrogénase
(G6PD) est aussi connue sous de nombreuses formes
rapportées à des séquences allèliques
différentes d'un même gène moléculaire
(voir exercice 4 p 38, Bordas 1èreS). Cette
enzyme intervient dans une voie secondaire de l'oxydation du glucose
(parallèle à la glycolyse) appelée voie des
pentoses phosphate qui produit en grande quantité des
transporteurs d'électrons et de protons (pouvoir
réducteur) réduits (voir cours de Terminale en
spécialité). . Elle catalyse, en présence d'ions
Mg2+ comme cofacteur, la réaction de
déshydrogénation du glucose-6-phosphate en
6-Phospho-glucono-è-lactone avec la transformation simultanée
du NADP+ (nicotine amide di nucléotide phosphate, un
transporteur d'électrons et de protons), deuxième
substrat, en NADPH+H+.
Remarque:
S'il est de bonne guerre qu'un laboratoire pharmaceutique (Roche) -
qui a investi beaucoup (mais vraiment beaucoup) d'argent (et pas
seulement de l'argent mais aussi en quelque sorte son image) dans la
génomique - distribue gratuitement un CD-Rom (superbe -
http://www.rochegenetics.com
- Programme de formation en génétique) très
orienté sur les avantages que l'on a à continuer la
recherche génétique, le rôle d'un enseignant me
semble être d'essayer de prendre du recul. Entre les
déclarations d'intention, les hypothèses et les
idéologies, politiques ou autres, il faut faire le tri. Ce ne
sont pas les étudiants qu'il faut convaincre par un cours
orienté ce sont les enseignants. J'ai assez confiance dans
leur capacité à prendre du recul, surtout au bout de
quelques années de carrière. Ce n'est pas de la
frilosité, c'est de la sagesse. Une fois encore le travail de
l'enseignant est, comme celui de tout pédagogue, à la
fois un encouragement à rêver, à inventer, et une
mise en garde basée sur l'expérience du passé
qui doit toujours être le support de ce que l'on construit, un
travail non pas dépassé mais assimilé et
intégré.
2.3.3 Retour à la cellule et au travail du vivant
|
Une comparaison entre
un travail biologique et un
travail humain
|
- Globalement le rendement d'un travail humain est
déterminé par 3 paramètres:
- le nombre de travailleurs,
- la rentabilité individuelle de chaque
travailleur liée aux conditions internes et
externes à celui-ci
- et enfin à la disponibilité des
matériaux et de l'énergie ou encore de
l'information nécessaire au travail.
- Pour les système enzymatiques nous pouvons
penser que ces mêmes paramètres
interviennent:
- le nombre de complexes enzymatiques par exemple dans
une cellule dépend du niveau d'expression des
gènes associés aux enzymes de ce complexe
(et de la fidélité du mécanisme de
l'expression de l'information génétique);
le contrôle est principalement
génétique mais le niveau de l'expression de
l'information génétique dépend
étroitement des conditions cytoplasmiques et
environnementales;
- l'activité individuelle de chaque complexe
dépend des conditions (pH, T,...) dans lequel
chacun se trouve; et les conditions peuvent être
très différentes d'un complexe à
l'autre; le contrôle est double: à la fois
cytoplasmique et environnemental; le contrôle
génétique est ici minimal, même si,
en dernier ressort, il peut faire intervenir des
protéines régulatrices, associés
à des gènes de contrôle
|
L'information
génétique au cœur de la vie.
Un schéma plus complexe que celui donné
précédemment (voir cours
de seconde ch1.2 par exemple ou
directement le schéma
simplifié), mais plus exact
et montrant les interactions entre les informations
génétiques, cytoplasmiques et environnementales. On
notera que l'information génétique (ADN et ARNm)
intervient dans la vie cellulaire par l'intermédiaire de ses
produits (autres ARN et peptides). Les produits de l'information
génétique ne font pas vraiment partie de l'information
génétique mais peuvent appartenir à
l'information cytoplasmique ou environnementale selon leur
destinée dans la cellule (notée ici
participation). Un produit de l'information
génétique peut exercer un
rétrocontrôle sur l'expression de sa propre
information génétique c'est-à-dire celle dont il
est l'expression. On distingue les
rétrocontrôles transcriptionnels (exercés sur la
transcription) et post-transcriptionnels (exercés sur la
maturation des ARN et la traduction). Quand on dit qu'un
gène module l'activité d'un autre gène, c'est en
fait des produits de deux informations génétiques qui
interagissent avec leur information génétique
réciproque. L'information génétique (ADN
et ARNm) ne peut être que répliquée,
transmise, réprimée ou exprimée; toutes ces
étapes étant contrôlées par d'autres
informations cellulaires. Il est dérisoire de désigner
par le terme d'épigénétique*
toute information non génétique. L'utilisation de ce
terme enferme dans une vision "moléculariste" de la cellule
à laquelle nous préférons une vision
biologique.
Remarques:
- Des substances qui ne sont pas sous un contrôle
génétique direct peuvent modifier la transcription
ou la traduction. Un article récent par exemple (Pascale
Fanen: «Un traitement pharmacologique restaure le
gène déficient», La
Recherche, 370, décembre 2003, 26-27 M. Wischanski et
al., NEJM, 349, 1433, 2003, résumé
sur la page de l'ancien
cours de spécialité) met en
évidence le rôle de certains antibiotiques; on sait
que de nombreux antibiotiques (et toxines) peuvent intervenir
directement dans différentes étapes de la
synthèse des protéines (voir
Biochimie, lehninger, p 927); la plupart du temps ce sont
des inhibiteurs (par exemple la puromycine qui se lie à un
aa comme un tRNA mais bloque la translocation du ribosome, ce qui
conduit à une interruption prématurée de
l'élongation du peptide...). Mais il existe aussi des
antibiotiques (petits peptides, glucides...) qui peuvent au
contraire favoriser l'allongement de la chaîne peptidique
comme par exemple la gentamicyne qui est connue
comme les autres antibiotiques du groupe des aminoglycosides pour
pouvoir supprimer l'effet des codons stop intercalés dans
l'ARN avant le codon stop terminal normal. Ce rôle a
déjà été testé par exemple pour
les malades atteints de mucoviscidose qui présentent une
double (homozygote) ou simple (hétérozygote)
mutation du gène de la protéine CFTR induisant un
codon stop prématuré (2 à 5% seulement des
malades sont le gène CFTR est reconnu comme
présentant une mutation défavorable). Ils peuvent
ainsi être efficacement soignés par l'administration
nasale de l'antibiotique (pendant 2 semaines en
pulvérisation) et synthétiser alors des
protéines CFTR normales.
- Les molécules chaperons (qui
"chaperonnent" les autres protéines en les
protégeant des stress, notamment thermiques) forment un
nouveau groupe de protéines fonctionnelles
découvertes vers les années 1960 mais
dénommées ainsi depuis 1978. Ces protéines
empêchent par exemple les agrégats de
protéines associées par leurs groupements
hydrophobes formés à la suite d'un choc thermique
(température supérieure à 40°C). Par
exemple, chez Escherichia coli on connaît une
protéine chaperon nommée GRO E faiblement
synthétisée à 37°C (1% des
protéines de la bactérie) mais dont la
synthèse est fortement activée si l'on augmente la
température (10% de la masse protéinique à
46°C).
Chez la levure Saccharomyces cerevisiae, la drosophile et l'homme,
on a isolé un groupe de gènes (respectivement 9, 8
et 10) associés à des molécules chaperons
dites chaperonnes 70kDa (du fait de leur poids moléculaire;
le dalton (Da), qui est la masse d'un atome d'hydrogène,
vaut 1,67.10-24 g) intervenant dans le trafic
intracellulaire des protéines. Les gènes (ou du
moins les mécanismes de leur expression) sont
thermosensibles. Par exemple chez la levure, le gène SSA,
est transcrit à 23 °C, mais à
37 °C sa transcription est multipliée par cinq.
Le gène SSA2 est transcrit normalement à toute
température. Les gènes SSA3 et SSA4 sont transcrits
uniquement à 37 °C. Il faut bien voir que ce
n'est pas une variation de l'activité des enzymes
transcriptionnelles et traductionnelles qui est ici en cause mais
bien une modulation de l'expression de l'information
génétique par un facteur externe.