|
Sheldon Penman, 1995, Proc. Natl.
Acad. Sci. USA |
|||||||
|
Traduction personnelle mot-à-mot non garantie Le photos étant volontairement de
qualité réduite il sera nécessaire de
se reporter aux photos originales. |
|
|
|||||
|
|
|||||||
|
RÉSUMÉ La biologie cellulaire a longtemps travaillé avec une fausse idée de la structure cellulaire puisque les électromicrophotographies conventionnelles ne montrent pas la plupart des composants de l'architecture cellulaire. Ce n'est pas une faiblesse technique intrinsèque à l'imagerie électronique, puisque qu'une technique, récente, et très simple de microscopie électronique (ME) donne des images beaucoup plus précises et détaillées de la structure cellulaire. Cette revue explique brièvement le problème de ma ME conventionnelle et de quelle façon il est résolu. Des exemples d'images obtenues montrent qu'elles diffèrent nettement des images habituelles de l'architecture interne de la cellule. L'introduction d'un moyen plus précis pour visualiser la structure cellulaire arrive à point nommé étant donné que la perception du rôle de l'architecture cellulaire est en train d'évoluer rapidement. Je crois que la transformation conceptuelle qui en résultera se révélera conforme à la phrase de Kuhn si fraie et cependant galvaudée être un «changement de paradigme.» Une école de recherche nouvelle dépasse les sujets d'intérêt actuels, en grande partie fondés sur la biochimie des solutions, pour se focaliser sur la structure de la cellule, sa réalisation et son organisation et, plus important, sur rôles sociaux fondamentaux dans les fonctions biochimiques et développementales. Comme on l'expliquera ici, la ME peut intégralement et fidèlement figurer la structure de cellule, et mettre en place de nouveaux concepts, en apportant une aide à la recherche structurale. Malheureusement, en même temps, l'étude de la structure de la cellule a été délaissée comme n'ayant qu'un pur intérêt d'observation, par comparaison - défavorable - avec la biochimie réductionniste supposée plus rigoureuse. Même si un tel dénigrement n'a jamais été justifié, celui-ci est de plus en plus inapproprié et hors de propos; des études contemporaines sur la structure de la cellule sont tout aussi rigoureuses et réductionnistes que d'autres. En outre, l'architecture cellulaire s'avère critique pour la compréhension de nombreuses fonctions des cellules différenciées (1-3), de la forme cellulaire (4), du contrôle de la croissance cellulaire, de la transformation néoplasique , et d'un large éventail d'événements de régulation (5, 6). De telles études rigoureuses ajoutent un aspect mécanique et physique au côté chimique sur lequel on insiste davantage habituellement. Seule une telle recherche peut découvrir les mécanismes de coopération entre cellules qui construisent des modèles de tissus et la forme de l'organisme, qui sont des sujets récalcitrants à l'approche biochimique. Enfin, la recherche structurale mettra en lumière les casse-têtes les plus frustrants et réfractaires : la nature et la localisation des instructions génomiques dictant la forme des cellules, des tissus et, en fin de compte, des organismes. Un obstacle majeur à l'étude de la structure a été l'incompréhensible sentiment selon lequel elle n'existait tout simplement pas. Les électronographies classiques de cellules montrent des cellules avec guère plus de structure qu'un marshmallow. L'intérieur en grande partie sans relief a fortement découragé la recherche d'éléments qui seraient sous le contrôle du génome et qui détermineraient la forme de la cellule, des tissus et de l'organisme. Malgré son utilisation universelle, l'image au ME conventionnel fut considérée comme fortement incomplète. Paradoxalement, la microscopie optique permet de montrer une architecture du cytosquelette souvent extraordinaire (7-9), mais avec une résolution relativement faible. Le ME, bien plus puissant ne pourrait montrer l'intérieur de la cellule que comme un plasma sans forme où les organites seraient apparemment dispersés. Il n'y apparaît aucun cadre sur lequel reposerait la forme des cellules, leur polarité et l'évidente distribution ordonnée des organites. Le noyau n'a montré aucun échafaudage ou matrice qui expliquerait l'organisation des longueurs énormes de chromatine. En outre, la chromatine apparaît comme des points plutôt que sous forme des solénoïdes épais que l'on supposait. La mitose semble être un processus largement inexplicable, presque magique. Néanmoins, malgré les inquiétudes sur leur exactitude, les électronographies de sections fines incluses ont continué d'être la norme. Elles montrent très bien certaines choses comme les sections perpendiculaires de membranes et de l'intérieur d'organites. L'incapacité à montrer la structure interne des organites n'est pas un défaut du ME. L'image "marshmallow" est entièrement due à l'inclusion dans du plastique que l'on utilise pour les échantillons observés par ME traditionnelle afin de permettre l'obtention de sections ultrafines. La section d'échantillons inclus dans la résine trouve son origine dans les débuts de la ME lorsque les résultats étaient hasardeux et que les cellules étaient considérées comme extrêmement fragiles. Aujourd'hui, l'inclusion est souvent un obstacle à la compréhension. La ME d'échantillons non inclus révèle l'intérieur de la cellule comprenant un étonnant réseau de structures complexes. Ces réseaux dynamiques et structuraux sont présents dans tout le cytoplasme et les noyaux de toutes les cellules et, je crois, devrait être considérée comme le véritable "cytosquelette". Le terme cytosquelette
désigne souvent uniquement les trois types de
filaments que l'on observe habituellement par fluorescence
((microfilaments, filaments intermédiaires et
microtubules). Ces filaments ne sont qu'une petite partie de
la cytostructure nouvellement révélée
(peut-être 10-15% d'après les dosages
biochimiques). Enlever la
résine obscurcissante et les protéines
solubles permet d'obtenir des électronographies
étonnamment claires de l'armature de filaments dans
le cytoplasme et le noyau. La recherche sur la structure
cellulaire nécessite que cesse la lutte avec la
conception sans avenir de la cellule marschmallow.
|
|
A major impediment to the study of cell structure has been the understandable perception that there was simply not very much of it. The conventional electron micrograph of cells shows cells with little more of a framework than a marshmallow. The largely
featureless interior strongly discouraged searching for
organizing prin- ciples that, instructed by the genome,
determine cell, tissue, and organism form. Despite its
universality, the conventional EM image was realized as
seriously incomplete. Paradoxically, optical microscopy
could display elaborate, often dra- matic cytoskeletal
architecture (7-9) but at relatively low optical resolution.
The far more powerful EM could show the cell interior only
as formless "plasm" with organelles seemingly scattered
about. There appeared no cell framework for determining cell
shape, cell polarity, and the obviously nonrandom morphology
and distribution of organelles. The nu- cleus showed no
scaffold or matrix for organizing enormous lengths of
chroma- tin. Also, the chromatin appeared only as dots
rather than as the expected thick chromatin solenoids.
Mitosis seemed a largely inexplicable, almost magical pro-
cess. Nevertheless, despite concerns about accuracy, the
embedded thin-section elec- tron micrograph continued to be
the norm. They do show some things ex- tremely well-e.g.,
membrane profiles and cross-sections of organelle interiors.
Failure to image internal cell structure is not a fault of
the EM. The "marshmallow" image is entirely due to the
plastic embedding used in traditional EM sam- ples to allow
cutting ultrathin sections. The plastic-embedded section
originated in the earliest days of EM when its short-
comings were not obvious and cells were considered extremely
fragile. Today, embedding is often an obstacle to
understanding.
|
|||||
|
|
||||||
|
|
|||||||
|
Pourquoi la ME conventionnelle des sections d'éléments inclus dans la résine cache-t-elle la majorité de la structure interne de la cellule ? Une simple considération des principes élémentaire de l'optique montre pourquoi les habituelles sections incluses montrent si peu de la réalité. Toute microscopie exige qu'un échantillon diffère optiquement (ou électroniquement) de son environnement. Les microscopes optiques et ME forment tous deux une image agrandie en focalisant les perturbations des rayons éclairants. Les images d'un microscope à champ clair résultent de l'absorption différentielle de colorants sélectifs. De même, les instruments optiques sensibles à la phase utilisent des interférences pour construire une image de l'écart entre le retard de la lumière dans l'échantillon par rapport milieu environnent. De tels instruments interférentiels peuvent être particulièrement sensibles à de faibles variations des propriétés optiques de l'échantillon. Même si les manuels de biologie disent souvent le contraire de façon erronée, le ME est avant tout un Instrument de contraste de phase. Bien que n'étant pas souvent regardé ainsi, c'est un instrument extrêmement sensible qui peut facilement visualiser des objets translucides aux électrons, comme les fibres protéiques de petit calibre qui ne nécessitent aucune coloration par un métal lourd. L'extrême sensibilité et la résolution du ME sont complètement renversées par l'inclusion des échantillons dans la résine plastique qui avoisine leurs propriétés optiques aux électrons. La composition atomique de la matière plastique, qui détermine leur indice de réfraction aux électrons est presque identique à celle des échantillons biologiques. Pour un électron, l'échantillon est alors pratiquement homogène dans tout son volume et il ne reste peu de différence de phase entre l'échantillon et de son environnement qui puisse servir à former une image au ME. Toute personne qui a, par inadvertance, essayé de voir une section sans coloration préalable sait combien l'écran reste blanc. La production d'une image avec inclusion de l'échantillon exige la "coloration" de la section afin que les atomes de métaux lourds se lient à des à des parties de l'échantillon faisant saillie au-dessus de la surface de la section. Seuls ces atomes de surface donnent l'image familière du ME; rien de ce qui se trouve à l'intérieur n'est visualisé. La plupart des organites et des fibres se trouvent être en dessous de la surface de la section où ils restent invisibles. Les filaments en particulier sont difficiles à visualiser, car ils doivent se trouver dans le plan de section, car sinon ils apparaissent comme de simples points. En dehors de l'image des zones densément colorées des organites et des membranes pénétrant la surface de la section, l'intérieur de la cellule apparaît quasiment vide de toute forme. Cette technique d'inclusion plastique dense pourrait nous sembler aujourd'hui curieuse, mais il y a 40 ans, elle a servi à donner un sens aux images qui autrement apparaissent totalement confuses du fait de l'apparence inattendue des protéines solubles. Les sections montraient très bien les membranes, qui sont alors devenues d'un intérêt primordial. Le prix exigé pour cette fausse clarté était le masquage de ce nous allons considérons maintenant comme étant la structure interne essentielle des cellules. Il y a 15 ans, Keith Porter (10) réintroduit une technique beaucoup plus ancienne de ME, qui monte des assemblages de cellules sans matériau d'inclusion. Les images résultantes présentaient un réseau dense, complexe et polymorphe qu'il appela le "réseau microtrabéculaire." Les images semblaient mystérieuses et ne pouvaient être reliés à rien de ce que l'on trouvait dans les microphotographies orthodoxes ; le terme «artefact», utilisé dans son sens péjoratif, a été utilisé. La répétition des études de Porter a d'abord été difficile, car peu de chercheurs avaient accès au 1-MeV-ME nécessaire. La haute tension a permis de pénétrer le dense treillis microtrabéculaire. Cependant, un simple développement technique a rendu le microscope à haute tension inutile ; les sections ultra-fines, sans résine, permettent de voir les cellules avec des microscopes très répandus de 80 kV et d'obtenir des résultats équivalents à ceux de Porter. Les sections sans résine sont faites par inclusion de l'échantillon dans un milieu provisoire et en coupant des sections avec un ultramicrotome. Les coupes ultrafines obtenues sont placées sur grille, traitées avec un solvant pour enlever le milieu d'inclusion temporaire, et ensuite séchées au point critique du CO2. Les échantillons fixés au Glutaraldehyde, même lorsqu'ils sont intensément traités, sont remarquablement résistants et semblent garder leur forme même lorsqu'ils subissent d'importantes manipulations. La technique de la section sans résine fût d'abord développée par Wolosewick (11) en utilisant du polyéthylène glycol (PEG) pour l'inclusion provisoire. Nous avons trouvé l'utilisation du PEG techniquement difficile et nous avons développé une technique alternative basée sur l'utilisation de diéthylène distéarate (DGD) pour l'inclusion provisoire (12). La plupart trouvent que les coupes au DGD sont plus faciles qu'au PEG étant donné qu'il n'est pas soluble dans l'eau et qu'on peut donc les faire flotter dans l'eau qui remplit habituellement le bac du microtome. Récemment, nous avons adopté une forme modifiée du DGD (Antibed; fourni aimablement par la EMCorp, Chestnut Hill, MA) qui permet d'éviter certains problèmes avec brillance. |
|
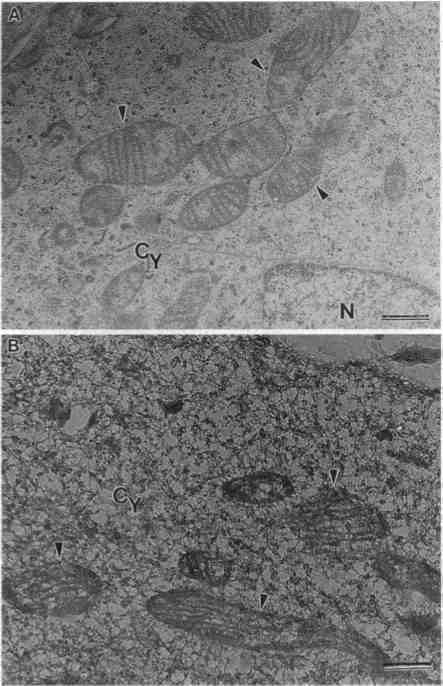
Why the Conventional Embedded EM Section Conceals Most of the Cell's Interior Structure A brief reflection on elementary optical principles shows why the usual embedded section shows so little of reality. All mi- croscopy requires that a sample differ optically (or electron optically) from its surroundings. Both light microscopes and EMs form a magnified image by focusing perturbations in the illuminating rays. The images in a bright-field microscope result from differential absorption affected by selective stains. Similarly, phase-sensitive optical instruments use interference to image the differential retardation of light by the sample compared with the sur- rounding milieu. Such interference instru- ments can be quite sensitive to subtle optical properties of the sample. Although biology textbooks often incorrectly state otherwise, the EM is primarily a phase- contrast instrument. Although not often thought so, it is an extremely sensitive instrument that can easily image relatively electron lucent objects, such as small cal- iber protein fibers, and needs no heavy metal staining. The extreme sensitivity and resolution of the EM are completely subverted by em- bedding samples in a plastic resin that matches their electron optical properties. The atomic composition of plastics, which determines their electron index of refrac- tion, is almost identical to that of biological samples. To an electron, the sample volume is now essentially homogeneous and it ex- periences little phase difference between the sample and its surroundings that could serve to form an EM image. Anyone who has, by oversight, tried to view an EM sec- tion without poststaining knows how blank the viewing screen will be. Producing an image with an embedded sample requires "staining" the section so that heavy metal atoms bind to bits of sample protruding through the section surface. Only these surface atoms form the familiar EM image; nothing in the section interior is portrayed. Most or- ganelles and fibers lie below the actual section surface where they remain invisi- ble. Filaments in particular are difficult to image since they must lie in the plane of the section surface or else appear as a mere dot. Apart from the image of stain bound to those dense organelles and membranes penetrating the section sur- face, the cell interior appears largely form- less. This technique of dense plastic em- bedding might strike us today as curious, but 40 years ago it served to make some sense from images otherwise hopelessly confused by the unexpected appearance of soluble proteins. The sections showed membranes very well, which were then of primary interest. The price exacted for this seeming clarity was the masking of what we now consider critical internal cell structures. Embedment-free EM About 15 years ago, Keith Porter (10) reintroduced a much older EM technique, one that viewed whole mounts of cells without any embedding material. The re- sulting images showed a dense, complex, polymorphic mesh that he termed the "microtrabecular lattice." The images seemed mysterious and could not be re- lated to anything in orthodox micro- graphs; "artifact," used in its pejorative sense, was heard. Repeating Porter's stud- ies was initially difficult since few had access to the necessary 1-MeV EM. The high voltage served to penetrate the dense microtrabecular lattice. However, a sim- ple technical development has made a high-voltage microscope unnecessary; the ultrathin, resinless section allows viewing cells with a garden variety 80-kV micro- scope and achieves results equivalent to those of Porter. Resinless sections are made by embed- ding the sample in a temporary medium and FIG. 1. Comparison of transmission electron micrographs of whole HeLa cell sections prepared through the conventional Epon technique (A) and the DGD resinless method (B). (A) Mitochondria (arrowheads) are seen "floating" in the cytoplasm (Cy) of a HeLa cell that was embedded in Epon, sectioned, and conventionally poststained. N, nucleus. (B) Resinless section of a fixed HeLa cell that was initially embedded in DGD shows mitochondria (arrowheads) enmeshed in the cytoplasmic microtrabecular lattice that is revealed through this technique. (Bar = 100 nm.) 5252 Review: Penman Proc. Natl. Acad. Sci. USA 92 (1995) 5253 cutting sections on the ultramicrotome. The resulting ultrathin sections are placed on a grid, extracted with solvent to remove the temporary embedding medium, and then dried through the CO2 critical point. Glu- taraldehyde-fixed samples, even when ex- tensively extracted, are remarkably tough and appear to maintain their form even when undergoing considerable manipula- tion. The resinless section technique was first developed by Wolosewick (11) using polyethylene glycol (PEG) as the provi- sional embedment. We found using PEG technically difficult and developed an alter- native technology based on using diethylene glycol distearate (DGD) for interim embed- ding (12). Most find the DGD cuts far more easily than PEG and, since it is not water soluble, it can be floated in the usual water- filled microtome trough. Recently, we have adopted a modified form of DGD (Antibed; the kind gift of the EMCorp, Chestnut Hill, MA) that avoids some problems with brit- tleness. |
|||||
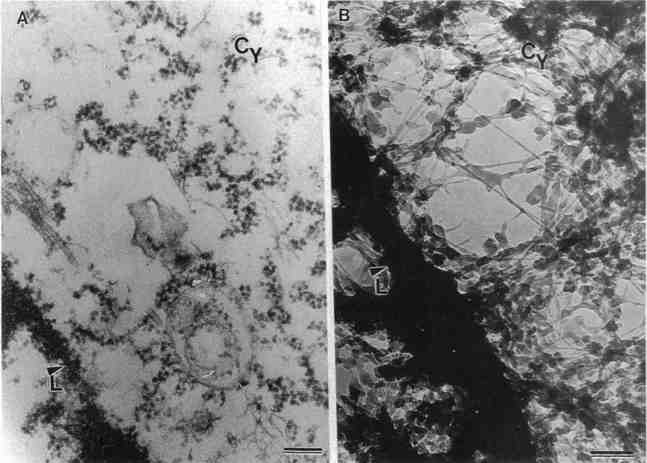 Fig. 2. Préparations du cytosquelette de cellules HeLa vus par Microscopie Electronique (ME) classique (A) et non incluse (B). La section sans résine montre, par contraste avec celle obtenue classiquement, les filaments du cytosquelette interconnectés qui s'étendent à travers le cytoplasme (Cy) et sont ancrés sur la lamina nucléaire (barre = 100 nm). (voir commentaire dans le texte ci-dessous où les polyribosomes sont décrits) |
|
||||||
|
|
|||||||
|
Face aux protéines solubles désormais visibles L'obtention de sections sans inclusion n'est que la première étape du dévoilement de la cytoarchitecture. Maintenant, les protéines solubles, convenablement cachées à l'intérieur des sections avec inclusion pendant un demi-siècle, sont visibles. Le contraste avec les micrographies conventionnelles est frappant, mais peu informatif. La Fig. 1A montre le cytoplasme d'une cellule HeLa incluse dans l'habituel Epon avec une précoloration de la section, alors que la Fig. 1B montre une section du même matériel cellulaire sans coloration ni inclusion. Le réseau trabéculaire fantomatique visible en 1B et présent en 1A, mais invisible est dû au réseau de protéines solubles réticulées fixées au sein d'une structure stable par le fixateur. Les protéines solubles représentent la majeure partie de la masse cellulaire et cachent les organites, comme les mitochondries, qui ne peuvent qu'être entrevues. Leur présence a sûrement découragé les premiers microscopistes, car, une fois visibles, elles cachent tout le reste. Le problème des protéines solubles a très probablement conduit à l'adoption de la technique des inclusions qui au moins montrait les limites des membranes. Grâce à notre connaissance accrue de la structure cellulaire, nous pouvons maintenant faire beaucoup mieux qu'ensevelir les protéines solubles dans de la résine, cachant ainsi la majeure partie de l'intérieur de la cellule. Au lieu de cela, nous pouvons enlever sélectivement et totalement les protéines solubles par extraction avec un détergent non ionique. Le mécanisme de l'extraction par le détergent est souvent mal compris ; les détergents anioniques (par opposition aux détergents ioniques) n'affectent pas les protéines et donc ils "n'extraient" rien au sens habituel de ce terme. Au lieu de cela, le détergent dissous simplement les phospholipides membranaires, en supprimant la barrière de la membrane plasmique et permet alors aux protéines solubles de diffuser passivement à l'extérieur. Si le solvant d'extraction est tamponné à un pH voisin de celui de l'intérieur de la cellule , le procédé laisse les structures cytosquelettiques intactes et avec une morphologie presque primitive. Le cytosquelette après extraction au détergent, libre de protéines solubles, est potentiellement idéal pour les études biochimiques et au ME. Cependant, la ME conventionnelle utilisant des sections avec inclusion est particulièrement stérile pour visualiser le cytosquelette après extraction au détergent. Les réseaux en 3 dimensions des filaments du cytosquelette sont précisément le type de matériel qui donne les images les moins riches dans les sections classiques. La figure 2A montre la faible image des structures cytosquelettiques dans une section après action du détergent et inclusion à l'Epon. Des parties d'une région cytoplasmique (en haut à droite) ainsi que du noyau (en bas à gauche) sont visibles. Les ribosomes sont les composants les plus visibles du cytoplasme dû à l'affinité de leurs ARN pour les ions uranyle du colorant. Les groupes non aléatoires visibles des polyribosomes reflètent leur liaison au cytosquelette qui, bien sûr, n'est pas visible ici. Certains ribosomes sont associés avec ce qui pourrait être des restes du réticulum endoplasmique rugueux. L'image du noyau est également sans information, montrant la chromatine,principalement agglutinée, et presque rien d'autre. La figure 2B montre l'image étonnante et fortement contrastée du cytosquelette obtenue par une section sans résine ni colorant. Le réseau microtrabéculaire de protéines solubles a bien sûr, disparu, permettant d'avoir une vue dégagée du cytosquelette. La profusion des structures du cytosquelette comprend des microfilaments et des filaments intermédiaires, des polyribosomes fixés au niveau du cytosquelette, et une myriade de petites structures ne sont pas encore identifiées. L'espace nucléaire montre des agglomérats de fibres de chromatine épaisse et des fibres minces, inédites, qui sont les filaments de la matrice nucléaire. La simple et efficace extraction par des tensioactifs non ioniques révèle une cytostructure hautement intriquée qui, contrairement à cellule non structurée, semble appropriée pour véhiculer des instructions architecturales complexes. Les sections sans résine sont des objets tridimensionnels et donnent de vraies images stéréoscopiques lorsqu'on les regarde avec un décalage visuel. En trois dimensions, les réseaux cellulaires ressemblent à des structures de "tenségrité" (???) c'est-à-dire, construit à partir d'éléments subissant des tractions et compressions, mais pas de courbure. Toutefois, l'extraction par le détergent a un sérieux revers : la solubilisation de toutes les membranes cellulaires. Nous verrons plus loin que l'extraction saponine qui enlève sélectivement le cholestérol des membranes, en laissant une grande partie des phospholipides, préserve une partie significative des membranes internes de la cellule. |
Coping with the Newly
Visible Soluble Proteins |
||||||
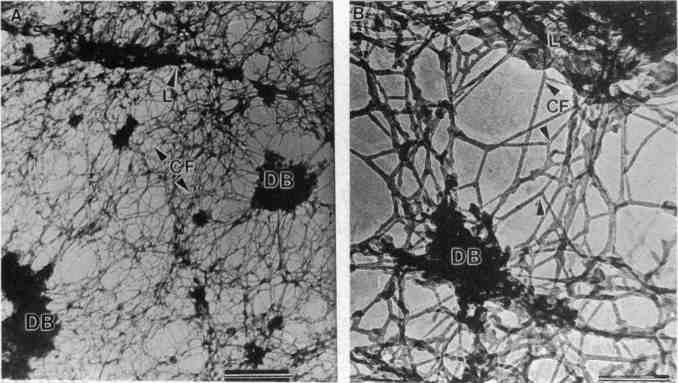 Fig. 3. Ultrastructure sans inclusion des core filaments (non traduit ; filaments fondamentaux = filaments de cœur) de la matrice nucléaire. Les micrographies montrent à faible (A) et fort (B) grossissement les connexions entre les composantes de la matrice nucléaire. Le réseau anastomosé des core filaments (CF) est entremêlé d'amas denses aux électrons qui forment les corps denses (DB) et se connecte avec la lamina nucléaire (L). (A, barre = 500 nm;. B, barre = 200 nm) (voir commentaire de la figure dans le texte) |
|
||||||
|
|
|||||||
|
Les "core filaments" de la matrice nucléaire L'extraction sélective peut faire davantage que de retirer les protéines solubles. Avec des modifications convenables, l'extraction peut être utile pour décortiquer progressivement les structures cellulaires, afin de pénétrer plus profondément dans l'intérieur de la cellule. Ce qui a été particulièrement utile dans la résolution des controverses qui entourent l'existence de la matrice nucléaire. Peu de sujets biologiques ont donné lieu ces derniers temps à un tel conflit passionné. Savoir exactement pourquoi l'idée d'une structure nucléaire nonchromatinienne a engendré un tel débat restera probablement une énigme. L'existence d'un échafaudage organisateur aurait sûrement pu être déduit de la complexité de l'organisation et de la dynamique nucléaire dont des composants solubles peinent à rendre compte. Très probablement, l'invisibilité complète de la supposée matrice nucléaire dans les micrographies d'alors, même après une réduction de la chromatine, a contribué à renforcer les doutes quant à sa réalité (13). La matrice nucléaire est cachée sous d'énormes quantités de chromatine dense aux électrons qui doit d'abord être enlevée avant une microscopie ou une analyse biochimique. Berezney et Coffey (14, 15) ont d'abord détecté biochimiquement la matrice nucléaire en tant que matériel nucléaire résiduel après une vigoureuse extraction de la chromatine à l'aide de DNase et de sel. Cette préparation et d'autres similaires ont servi dans de nombreuses et importantes recherches et ont permis d'établir la participation de la matrice à des événements nucléaires majeurs (16-23). Les préparations étaient particulièrement utiles dans les premières études sur des récepteurs hormonaux spécifiques (18, 25, 26). Toutefois, la matrice nucléaire est restée invisible dans les micrographies électroniques classiques, provoquant le scepticisme sur son existence. Bien sûr, l'invisibilité était aussi un grave obstacle à une recherche plus poussée. Une section d'une inclusion au ME ne permet pas de voir la matrice nucléaire pour la même raison qu'elle ne permet pas de voir le cytosquelette; des sections sans résine sont bien meilleures dans ce but. La nécessité d'éliminer l'écrasante quantité de chromatine opaque aux électrons est une complication supplémentaire. La chromatine se lie fortement à la matrice nucléaire et la retirer nécessite des procédés relativement musclés. Réaliser l'élimination complète de la chromatine tout en minimisant la distorsion de la matrice a été un objectif technique de longue haleine. La Fig. 3 montre des images de sections sans résine de nos plus récentes préparations qui présente d'après nous la forme la plus native de la matrice à ce jour. La figure 3A montre le
réseau de filaments de la matrice nucléaire
alors que la Fig. 3B est un agrandissement de la
précédente. La matrice est constituée
d'un réseau dense et anastomosé de filaments
de 10 à 13 nm de diamètre. Nous avons
suggéré qu'ils soient nommés "core
filaments" car ils sont au centre de la plupart des
structures de la cellule. Bien qu'ils ressemblent
superficiellement à des filaments
intermédiaires, ils ne partagent aucun épitope
avec soit les lamines soit les filaments
intermédiaires et ne leur semblent donc pas
liés. Empêtré dans les filaments, il
existe de nombreux corps denses, dont certains sont riches
en composants intervenant dans l'épissage des ARN.
L'identification, la localisation, et
la caractérisation des composants de la matrice
nucléaire ne font que commencer. |
|
The Core Filaments of
the Nuclear Matrix |
|||||
|
|
||||||
|
|
|||||||
|
La structure de la cellule lors de la mitose Les réarrangements au cours de la division cellulaire sont sans doute au plus profond de la mémoire cellulaire. Alors que la biochimie spécifique à la mitose a constitué un domaine actif et fructueux de la recherche, peu de choses ont été présentées au sujet de l'architecture cellulaire lors de la mitose. À part les études sur le fonctionnement des microtubules, les questions les plus importantes sur la cytostructure mitotique demeurent. Ce casse-tête comprend les mystérieux agents qui effectuent les mouvements des chromosomes avant que ne se forme le fuseau, la machinerie qui dirige les fibres du fuseau précisément vers les kinétochores, les mécanismes de la dissociation du noyau ainsi que de sa reformation, etc. La ME classique à l'aide de sections avec inclusion ne peut que suggérer la profonde réorganisation du cytosquelette et de la matrice nucléaire lors de la mitose. Lorsque la lamina nucléaire disparaît en fin de prophase, le cytopsquelette et la matrice nucléaire se mélangent au sein d'une nouvelle structure qui occupe toute la cellule, mais qui reste jusqu'à présent en général invisible. Au mieux, les sections avec inclusion suggèrent seulement quelques composants de grande taille de l'appareil mitotique tels que les chromosomes, de courts segments de fibres du fuseau, et le centriole à l'occasion. La section sans résine rend visible un monde nouveau dans la cytostructure mitotique. Seules les protéines solubles ont besoin d'être extraites; il n'y a plus besoin d'extraire la chromatine puisqu'elle a été compactée en chromosomes. L'espace restreint empêche une présentation complète ici, mais la plaque métaphasique de la Fig.5 montre les informations uniques que la technique apporte. En comparaison avec les formes vagues et obscures suggérées par les micrographies classiques, les réseaux de filaments et de fibres du fuseau apparaissent ici comme net, bien défini, et beaucoup plus complexe que ce que l'on pensait. Un réseau de filaments complexe entremêle les chromosomes en métaphase de la Fig. 5, filaments qui sont la plupart du temps invisibles dans les micrographies électroniques classiques. Les faisceaux de filaments denses s'étendant horizontalement à partir des chromosomes sont les fibres du fuseau qui apparaissent dans leur intégralité, lorsqu'ils sont, comme ici, placés dans un plan d'une section épaisse comme ici. Les nombreux filaments fins sont particulièrement bien interconnectés entre les chromosomes et les fibres du fuseau. Ceux-ci peuvent provenir des core filaments de la cellule en interphase, mais une identification définitive nécessitera la mise au point d'anticorps spécifiques des core filaments. Le point majeur apporté par ces photos est que, plutôt que de flotter dans une mer amorphe, les principaux éléments de la cellule mitotique font partie d'un réseau squelettique complexe qui rend compte des événements mécaniques de la mitose.
|
|
Cell Structure at
Mitosis |
|||||
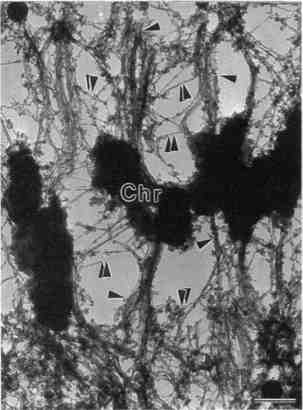 Fig. 5. Micrographie électronique sans résine d'une cellule HeLa mitotique. Les chromosomes (Chr) sont comme «suspendus» aux fibres du fuseau (pointes de flèches), qui sont reliées entre elles par le biais de nombreux filaments fins (flèches doubles). (barre = 500 nm). (voir commentaire de la figure dans le texte) |
|
||||||
|
|
|||||||
|
Les membranes dans les sections dans
résine : L'absence de membranes cellulaires est handicap issu des préparations des sections sans résine montrées précédemment. Ce n'est pas une faiblesse intrinsèque de la technique, mais le résultat de l'extraction de tous les phospholipides par le détergent utilisé pour éliminer les protéines solubles. La saponine, substituée au Triton X-100, perfore la membrane plasmique en extrayant préférentiellement le cholestérol (24). Ce détergent doux laisse une grande partie de la bicouche phospholipidique de la membrane plasmique intacte tout en permettant à des protéines solubles de diffuser à l'extérieur de la cellule. La Fig. 6 [ci-dessous] montre les mitochondries et d'autres organites délimités par une membrane empêtrés dans le cytosquelette dans des cellules ayant subi l'extraction à la saponine. Les crêtes mitochondriales apparaissent comme des zébrures sombres à l'intérieur de l'organite. Les filaments du cytosquelette sont en anastomose avec les surfaces de la membrane des organites. Cette image donne à penser que l'idée habituelle d'organites enveloppés d'une membrane comme des entités structurellement autonomes est incomplète. Les micrographies de sections non incluses dans la résine nous font savoir que les filaments du cytosquelette peuvent servir de cadre à l'organite qui est enveloppé par les bicouches lipidiques comme la toile de tente recouvre ses montants. Avec ce point de vue, le cytosquelette jouerait un rôle dans la structure des organites bien plus central que ce que l"on supposait. Un tel rôle pourrait expliquer les morphologies souvent complexes des membranes et organites.
|
|
Membranes in Resinless
Sections: |
|||||
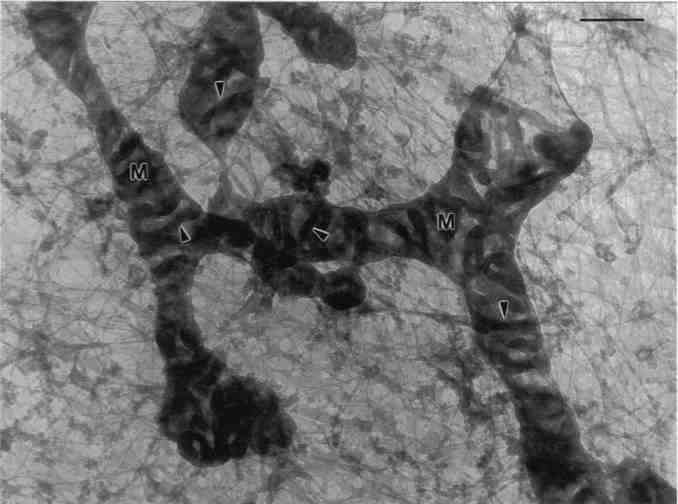 Fig. 6. Ultrastructure d'une cellule de muscle lisse de l'aorte de rat après extraction à la saponine. L'électronographie d'un montage de cellule complète montre des mitochondries (M) intimement connectées au réseau du cytosquelette. Les pointes des flèches désignent les crêtes mitochondriales (barre = 200 nm.) |
|
||||||
|
|
|||||||
|
Repenser la structure de la cellule La microscopie sans résine offre des images qui conduisent à des conclusions radicales. Tout d'abord, et c'est ce qui est probablement le plus important, la technique de section sans inclusion dans la résine peut être extrêmement puissante, mais seulement si elle est combinée avec la suppression du matériel obscurcissant. Aucune méthode n'est totalement satisfaisante dans tous les cas. Les protéines solubles, qui gênaient au début de la microscopie sans résine, sont plus facilement extraites avec un détergent non ionique, mais avec le défaut de perdre toutes les membranes internes. L'extraction avec la saponine a le même rôle tout en laissant beaucoup, mais pas toutes, les structures internes membranaires. La préparation à la saponine offre l'unique possibilité d'une étude plus approfondie des interactions membrane-cytosquelette. Enlever la chromatine si fortement liée tout en préservant une matrice nucléaire presque originale a été beaucoup plus difficile. Les préparations de matrice nucléaire avec les core filaments ont été la tentative la plus aboutie à de jour. Un deuxième point important est que les structures de la cellule sont remarquablement robustes. Elles conservent leur morphologie précise malgré l'extraction, l'inclusion et la désinclusion, la coupe et le séchage au point critique. Je soupçonne qu'une des raisons de l'adoption au départ de coupes incluses a été la croyance du côté éphémère des cellules une fois enlevées des tissus. L'inquiétude était sans doute justifiée dans les premiers temps avant que l'autolyse ne soit connue et contrôlée. Enfin, les techniques des sections sans résine sont particulièrement simples, la plupart des microscopistes les trouvent beaucoup moins difficiles à mettre en œuvre que les classiques sections ultra-fines à l'Epon. Il ne devrait pas y avoir d'obstacle technique à l'adoption de ces procédés pour ceux qui sont intéressés. Les images révélées par les techniques décrites ici contraignent à un sévère réexamen de ce que nous croyons être une cellule. L'ancienne vision, qui pourrait être désignée de façon simpliste comme le modèle "fluide",est celle d'un "plasma" modérément visqueux limité et contenu par une membrane plasmique constituée d'une bicouche lipidique dans laquelle les protéines sont insérées. La nouvelle idée est que le cytosquelette sert de cadre solide ou d'armature avec une limite externe d'une couche de protéines ou lamina où s'insèrent des lipides. Cette armature est ancrée sur une seconde couche protéique ou lamina qui est la surface du noyau, qui délimite une nouvelle armature, celle de la matrice nucléaire.  [petit schéma personnel de cette vision originale] Les armatures nucléaire et cytoplasmiques sont probablement conformes à la définition de la tenségrité (33), car la majorité des fibres biologiques sont résistantes à la tension et même à la compression alors qu'elles sont très peu résistantes à la courbure. Beaucoup de questions spécifiques du cytosquelette et de la fonction de la matrice nucléaire peuvent désormais être examinées à la lumière de notre meilleure compréhension de la structure cellulaire. En outre, des questions intellectuelles plus profondes sont posées par les réseaux structuraux complexes désormais connus dans les cellules; par exemple, où sont les instructions complexes qui organisent la mise en place de ces réseaux qui sous-tendent l'architecture ? Comme la matière noire en astronome, le codage d'une information sur l'architecture semble invisible et largement ignoré, bien que ses effets soient partout évidents. On pouvait ignorer ce sujet lorsque de nombreuses cellules semblaient ne pas avoir de structure. Une fois que nous avons ajouté un nouveau spectre de complexité structurale au codage du génome, nous ne pouvons plus ignorer que plus de 90 % du génome des vertébrés n'intervient pas directement dans la commande de la synthèse protéique. Cet ADN "supplémentaire" a longtemps été ennuyeux et apparemment sans utilité logique. Malgré sa fréquente et intense activité transcriptionnelle, il est souvent rejeté comme "poubelle", une désignation qui peut être intellectuellement apaisante, mais difficilement utile scientifiquement. La biologie moléculaire de la cellule ne peut vraiment atteindre une conception globale en ignorant la majorité de l'ADN d'un organisme. Forme et structure ne sont pas des sujets naturels pour la biochimie qui, dans le monde macroscopique, traite de quantités scalaires, c'est-à-dire de masses, taux, etc. Bâtir les concepts complexes aperçus dans tout texte d'anatomie ou de physiologie nécessite, à tout le moins, des instructions qui sont vectorielles; c'est-à-dire qui précisent le sens et le lieu. Ces instructions sont codées quelque part, et il semble très probable qu'elles se trouvent dans l'ADN fortement transcrit, mais "non codant pour les protéines". La construction incroyablement complexe d'organes, par exemple le cerveau ou les reins en ne spécifiant que les constituants protéiques (comme le suggère la formulation extrême de la biologie moléculaire selon laquelle les gènes sont tout simplement des protéines [???]) est peu probable. Une telle stratégie serait équivalente à essayer de décrire un pont ou un édifice en se contentant de donner une liste de pièces qui le composent. En effet, Gray's anatomy, vu avec l'oeil d'un ingénieur, suggère que la complexité des paquets d'instructions pour la morphologie d'un mammifère nécessite de vastes régions du génome : très probablement une grande part ou la majeure partie des 90 % (ou plus) du génome qui ne code pas pour les protéines actuellement ignorés. Je soupçonne que les biologistes cellulaires futurs seront émerveillés par la densité et l'ingéniosité des instructions du génome pour la structure tout en se demandant comment nous avons pu les ignorer pendant si longtemps.
|
|
Rethinking Cell
Structure |
|||||
|
1. Aggeler, J., Ward, J., Blackie, L. M.,
Barcellos-Hoff, M. H., Streuli, C. H. & Bissell, M. J.
(1991) J. Cell Sci. 99, 407-417. |
|
||||||
|
|
|||||||